Механизмы исправления ошибок во время репликации ДНК и ее репарация вследствие повреждений на протяжении всего жизненного цикла клетки.
Основные моменты:
-
Клетки имеют различные механизмы предотвращения возникновения мутаций – необратимых изменений в ДНК
-
В процессе синтеза ДНК, большинство ДНК-полимераз «проверяют свою работу» и проводят замену бо́льшей части ошибочно вставленных нуклеотидов. Этот процесс можно назвать исправлением ошибок.
-
Сразу после синтеза ДНК любые оставшиеся ошибочные нуклеотиды обнаруживаются и заменяются в так называемом процессе репарации ошибочно спаренных нуклеотидов.
-
Если ДНК повреждена, она может быть восстановлена с помощью различных механизмов, например, путём прямой репарации, эксцизионной репарации или путём восстановления двухцепочечных разрывов
- пострепликативной репарации.
Введение
Как ДНК связана с раком? Рак возникает при неконтролируемом делении клеток, когда игнорируются клеточные «стоп»-сигналы, что приводит к образованию опухоли. Это неправильное поведение клеток вызвано накопившимися мутациями — необратимыми изменениями последовательности ДНК клетки.
На самом деле, ошибки в процессе репликации и повреждения ДНК возникают в клетках нашего тела постоянно. Однако в большинстве случаев они не приводят к раку и даже не вызывают мутаций, такие ошибки обычно обнаруживаются и исправляются в процессе репарации ДНК. Если же повреждение исправить не удаётся, то в клетке включается механизм самоуничтожения — (апоптоз), который предотвращает передачу поврежденной ДНК дочерним клеткам.
Мутации возникают и передаются дочерним клеткам только тогда, когда эти механизмы не справляются. В частности, рак возникает в случае накопившихся в одной клетке мутаций генов, связанных с делением.
В этой статье мы подробно рассмотрим механизмы, используемые клетками для исправления ошибок, которые возникают в процессе репликации. К ним относятся:
-
Исправление ошибок – процесс, который возникает во время репликации ДНК.
-
Репарация ошибочно спаренных нуклеотидов, которая происходит сразу же после репликации ДНК.
-
Механизмы репарации, которые выявляют и исправляют повреждения ДНК на протяжении всего клеточного цикла
Исправление ошибок
ДНК-полимеразы — это ферменты, участвующие в репликации ДНК. Во время копирования ДНК большинство ДНК-полимераз «проверяют», корректный ли нуклеотид они добавляют. Этот процесс называется исправлением ошибок. Если полимераза обнаружит, что был добавлен неправильный нуклеотид, она сразу же удалит и заменит его и только после этого продолжит синтез ДНКstart superscript, 1, end superscript.
Репарация ошибочно спаренных нуклеотидов
Процесс исправления избавляет от основной массы ошибок, но не от всех. После создания новой ДНК запускается механизм репарации ошибочно спаренных нуклеотидов — удаления и замены ошибочно спаренных нуклеотидов, оставшихся в результате репликации. Исправление несоответствий между парами оснований также может включать в себя исправление небольших вставок и делеций, возникающих вследствие «соскальзывания» полимеразы с исходной цепи squared.
Как происходит восстановление неправильно спаренных нуклеотидов? Во-первых, белковый комплекс распознаёт неправильно спаренный нуклеотид и связывается с ним. Другой комплекс разрезает ДНК в области несовпадения, а ещё одна группа ферментов отщепляет некорректный нуклеотид вместе с небольшим участком вокруг него. Затем ДНК-полимераза заполняет этот пробел правильными нуклеотидами, а фермент ДНК-лигаза сшивает разрывы в цепиsquared.
Удивительно: как белки, участвующие в восстановлении ДНК, определяют, «кто прав» во время репарации ошибочно спаренных нуклеотидов? То есть, когда два основания неправильно соединены (как G (гуанин) и T (тимин) на рисунке выше), какое из этих двух оснований должно быть удалено и заменено?
У бактерий можно отличить исходную и дочернюю цепи ДНК по метилированным основаниям. На исходной цепи ДНК есть метильные (minus, start text, C, H, end text, start subscript, 3, end subscript) группы, присоединенные к некоторым из ее оснований, а у дочерней цепи таких групп еще нетcubed.
У эукариот процессы, позволяющие идентифицировать исходную цепь при устранении несоответствий, включают распознавание одноцепочечных разрывов, которые обнаруживаются только у дочерней цепи cubed.
Механизмы репарации ДНК
С ДНК может что-нибудь случиться практически в любой момент жизни клетки, а не только во время репликации. Фактически, ДНК постоянно повреждается из-за воздействия внешних факторов: ультрафиолетового излучения и радиации, химических веществ, не говоря уже о спонтанных процессах, которые протекают даже без вмешательства окружающей среды!start superscript, 4, end superscript
К счастью, наши клетки имеют механизмы восстановления, с помощью которых они находят и исправляют большинство повреждений ДНК. Можно выделить несколько типов репарации:
-
Прямая репарация. Некоторые повреждения ДНК, вызванные химическими реакциями, могут быть «исправлены» находящимися в клетке ферментами.
-
Эксцизионная репарация. Повреждение одного или нескольких нуклеотидов ДНК часто исправляется удалением и заменой поврежденного участка. При эксцизионной репарации оснований удаляется только поврежденное основание. В случае эксцизионной репарации нуклеотидов, как и в случае репарации ошибочно спаренных нуклеотидов, которое мы рассмотрели выше, удаляются целиком нуклеотиды.
-
Репарация двухцепочечных разрывов: Существуют два основных способа: негомологичное соединение концов и гомологичная рекомбинация. Они используются для восстановления двухцепочечных разрывов ДНК (когда вся хромосома разделяется на две части).
Прямая репарация
В некоторых случаях клетка может исправить повреждение ДНК, обратив вызвавшую его реакцию. Дело в том, что «повреждение ДНК» — это, как правило, присоединение к ней лишней группы в результате химической реакции.
Например, гуанин (G) может подвергаться реакции с присоединением метильной (minus, start text, C, H, end text, start subscript, 3, end subscript) группы к атому кислорода в азотистом основании. Если это не исправить, метил-содержащий гуанин будет связываться с тимином (Т), а не с цитозином (С) во время репликации ДНК. К счастью, у людей и многих других организмов есть фермент, который может удалить метильную группу, обратив реакцию, и тем самым вернуть азотистое основание в нормальное состояниеstart superscript, 5, end superscript.
Эксцизионная репарация оснований
Эксцизионная репарация оснований — это механизм, используемый для обнаружения и удаления определенных типов поврежденных азотистых оснований. Ключевую роль в нем играет группа ферментов, называемых гликозилазами. Каждая гликозилаза обнаруживает и удаляет определенный вид поврежденных оснований.
Например, в процессе реакции дезаминирования цитозин может превратиться в урацил — основание, обычно встречающееся только в РНК. Во время репликации ДНК урацил будет соединяться с аденином, а не с гуанином (в отличие от цитозина), поэтому такое превращение может привести к возникновению мутацииstart superscript, 5, end superscript.
Для предотвращения подобных изменений гликозилаза, являющаяся частью сигнального пути эксцизионной репарации, обнаруживает и удаляет дезаминированные цитозины. После того, как основание было удалено, удаляется и оставшаяся часть нуклеотида, а другие ферменты заполняют пробелstart superscript, 6, end superscript.
Эксцизионная репарация нуклеотидов
Эксцизионная репарация нуклеотидов — это еще один способ удаления и замены поврежденных оснований. В результате нее обнаруживаются и корректируются повреждения, которые искажают форму двойной спирали ДНК. Например, азотистые основания могут измениться, присоединив к себе громоздкие группы атомов, в частности, в результате воздействия химических веществ, содержащихся в сигаретном дымеstart superscript, 7, end superscript.
Эксцизионная репарация нуклеотидов также используется для устранения повреждений, вызванных ультрафиолетовым излучением, например, при получении солнечного ожога. Под воздействием УФ-излучения цитозин и тимин могут вступать в реакцию с соседними основаниями, которые также являются цитозином или тимином, образуя при этом связи, изменяющие форму двойной спирали и вызывающие ошибки в процессе репликации ДНК. Наиболее распространенный тип таких связей — тиминовый димер — он состоит из двух тиминовых оснований, вступающих в реакцию друг с другом и образующих химическую связьstart superscript, 8, end superscript.
При эксцизионной репарации нуклеотидов поврежденные нуклеотиды удаляются вместе с соседними нуклеотидами. В этом процессе хеликаза (фермент, раскручивающий ДНК) раскрывает ДНК, образуя пузырь, а ферменты, разрезающие ДНК, отсекают поврежденную часть пузыря. Полимераза заполняет пробел, а лигаза сшивает разрыв в цепиstart superscript, 9, end superscript.
Репарация двухцепочечных разрывов
Некоторые факторы окружающей среды, например, радиация, могут вызывать разрывы обеих цепочек ДНК (разделение хромосомы на две части). Такие повреждения ДНК, если верить комиксам, ведут к появлению супергероев, но могут встречаться и после реальных катастроф, например, Чернобыльской.
Двухцепочечные разрывы опасны, потому что большие сегменты хромосом и сотни содержащихся в них генов могут быть потеряны, если разрыв не будет восстановлен. Существует два способа восстановления двухцепочечных разрывов ДНК: негомологичное соединение концов и гомологичная рекомбинация.
При негомологичном соединении концов два разорванных конца хромосомы просто склеиваются обратно. Этот механизм восстановления является «грубым» и неточным, в результате в месте разрыва, как правило, либо теряются нуклеотиды, либо добавляются лишние, что может привести к мутациям. Но это в любом случае лучше потери целого фрагмента хромосомыstart superscript, 10, end superscript.
При гомологичной рекомбинации для восстановления разрыва используется фрагмент из гомологичной хромосомы, который соответствует поврежденной хромосоме (или из сестринской хроматиды, если ДНК была реплицирована). В этом процессе две хромосомы объединяются, и неповрежденная область гомологичной хромосомы или хроматиды используется в качестве матрицы для замены поврежденной области. Гомологичная рекомбинация работает «чище», точнее, чем негомологичное соединение концов, и обычно не приводит к образованию мутацийstart superscript, 11, end superscript.
Репарация ДНК и заболевания человека
Доказательства важности механизмов репарации получены на основе генетических заболеваний человека. Во многих случаях мутации в генах, которые кодируют белки, участвующие в репарации, связаны с наследственным раком. Например:
-
Наследственный неполипозный колоректальный рак (также называемый синдромом Линча) вызван мутациями в генах, кодирующих белки, которые участвуют в репарации ошибочно спаренных нуклеотидовstart superscript, 12, comma, 13, end superscript. Поскольку такие нуклеотиды не восстанавливаются, у людей, страдающих этим синдромом, мутации накапливаются гораздо быстрее, чем у здоровых. Это может привести к развитию опухолей толстой кишки.
-
Люди с пигментной ксеродермой очень чувствительны к ультрафиолетовому излучению. Это вызвано мутациями в белках, участвующих в эксцизионной репарации нуклеотидов. Когда они не функционируют, димеры тимина и другие виды повреждений, вызванные ультрафиолетовым излучением, перестают восстанавливаться. У людей с пигментной ксеродермой после нескольких минут пребывания на солнце могут возникнуть сильные солнечные ожоги, и около половины из них заболевают раком кожи в возрасте до 10 лет, если только они не избегают солнечных лучейstart superscript, 14, end superscript.
Механизмы исправления ошибок во время репликации ДНК и ее репарация вследствие повреждений на протяжении всего жизненного цикла клетки.
Основные моменты:
-
Клетки имеют различные механизмы предотвращения возникновения мутаций – необратимых изменений в ДНК
-
В процессе синтеза ДНК, большинство ДНК-полимераз «проверяют свою работу» и проводят замену бо́льшей части ошибочно вставленных нуклеотидов. Этот процесс можно назвать исправлением ошибок.
-
Сразу после синтеза ДНК любые оставшиеся ошибочные нуклеотиды обнаруживаются и заменяются в так называемом процессе репарации ошибочно спаренных нуклеотидов.
-
Если ДНК повреждена, она может быть восстановлена с помощью различных механизмов, например, путём прямой репарации, эксцизионной репарации или путём восстановления двухцепочечных разрывов
- пострепликативной репарации.
Введение
Как ДНК связана с раком? Рак возникает при неконтролируемом делении клеток, когда игнорируются клеточные «стоп»-сигналы, что приводит к образованию опухоли. Это неправильное поведение клеток вызвано накопившимися мутациями — необратимыми изменениями последовательности ДНК клетки.
На самом деле, ошибки в процессе репликации и повреждения ДНК возникают в клетках нашего тела постоянно. Однако в большинстве случаев они не приводят к раку и даже не вызывают мутаций, такие ошибки обычно обнаруживаются и исправляются в процессе репарации ДНК. Если же повреждение исправить не удаётся, то в клетке включается механизм самоуничтожения — (апоптоз), который предотвращает передачу поврежденной ДНК дочерним клеткам.
Мутации возникают и передаются дочерним клеткам только тогда, когда эти механизмы не справляются. В частности, рак возникает в случае накопившихся в одной клетке мутаций генов, связанных с делением.
В этой статье мы подробно рассмотрим механизмы, используемые клетками для исправления ошибок, которые возникают в процессе репликации. К ним относятся:
-
Исправление ошибок – процесс, который возникает во время репликации ДНК.
-
Репарация ошибочно спаренных нуклеотидов, которая происходит сразу же после репликации ДНК.
-
Механизмы репарации, которые выявляют и исправляют повреждения ДНК на протяжении всего клеточного цикла
Исправление ошибок
ДНК-полимеразы — это ферменты, участвующие в репликации ДНК. Во время копирования ДНК большинство ДНК-полимераз «проверяют», корректный ли нуклеотид они добавляют. Этот процесс называется исправлением ошибок. Если полимераза обнаружит, что был добавлен неправильный нуклеотид, она сразу же удалит и заменит его и только после этого продолжит синтез ДНКstart superscript, 1, end superscript.
Репарация ошибочно спаренных нуклеотидов
Процесс исправления избавляет от основной массы ошибок, но не от всех. После создания новой ДНК запускается механизм репарации ошибочно спаренных нуклеотидов — удаления и замены ошибочно спаренных нуклеотидов, оставшихся в результате репликации. Исправление несоответствий между парами оснований также может включать в себя исправление небольших вставок и делеций, возникающих вследствие «соскальзывания» полимеразы с исходной цепи squared.
Как происходит восстановление неправильно спаренных нуклеотидов? Во-первых, белковый комплекс распознаёт неправильно спаренный нуклеотид и связывается с ним. Другой комплекс разрезает ДНК в области несовпадения, а ещё одна группа ферментов отщепляет некорректный нуклеотид вместе с небольшим участком вокруг него. Затем ДНК-полимераза заполняет этот пробел правильными нуклеотидами, а фермент ДНК-лигаза сшивает разрывы в цепиsquared.
Удивительно: как белки, участвующие в восстановлении ДНК, определяют, «кто прав» во время репарации ошибочно спаренных нуклеотидов? То есть, когда два основания неправильно соединены (как G (гуанин) и T (тимин) на рисунке выше), какое из этих двух оснований должно быть удалено и заменено?
У бактерий можно отличить исходную и дочернюю цепи ДНК по метилированным основаниям. На исходной цепи ДНК есть метильные (minus, start text, C, H, end text, start subscript, 3, end subscript) группы, присоединенные к некоторым из ее оснований, а у дочерней цепи таких групп еще нетcubed.
У эукариот процессы, позволяющие идентифицировать исходную цепь при устранении несоответствий, включают распознавание одноцепочечных разрывов, которые обнаруживаются только у дочерней цепи cubed.
Механизмы репарации ДНК
С ДНК может что-нибудь случиться практически в любой момент жизни клетки, а не только во время репликации. Фактически, ДНК постоянно повреждается из-за воздействия внешних факторов: ультрафиолетового излучения и радиации, химических веществ, не говоря уже о спонтанных процессах, которые протекают даже без вмешательства окружающей среды!start superscript, 4, end superscript
К счастью, наши клетки имеют механизмы восстановления, с помощью которых они находят и исправляют большинство повреждений ДНК. Можно выделить несколько типов репарации:
-
Прямая репарация. Некоторые повреждения ДНК, вызванные химическими реакциями, могут быть «исправлены» находящимися в клетке ферментами.
-
Эксцизионная репарация. Повреждение одного или нескольких нуклеотидов ДНК часто исправляется удалением и заменой поврежденного участка. При эксцизионной репарации оснований удаляется только поврежденное основание. В случае эксцизионной репарации нуклеотидов, как и в случае репарации ошибочно спаренных нуклеотидов, которое мы рассмотрели выше, удаляются целиком нуклеотиды.
-
Репарация двухцепочечных разрывов: Существуют два основных способа: негомологичное соединение концов и гомологичная рекомбинация. Они используются для восстановления двухцепочечных разрывов ДНК (когда вся хромосома разделяется на две части).
Прямая репарация
В некоторых случаях клетка может исправить повреждение ДНК, обратив вызвавшую его реакцию. Дело в том, что «повреждение ДНК» — это, как правило, присоединение к ней лишней группы в результате химической реакции.
Например, гуанин (G) может подвергаться реакции с присоединением метильной (minus, start text, C, H, end text, start subscript, 3, end subscript) группы к атому кислорода в азотистом основании. Если это не исправить, метил-содержащий гуанин будет связываться с тимином (Т), а не с цитозином (С) во время репликации ДНК. К счастью, у людей и многих других организмов есть фермент, который может удалить метильную группу, обратив реакцию, и тем самым вернуть азотистое основание в нормальное состояниеstart superscript, 5, end superscript.
Эксцизионная репарация оснований
Эксцизионная репарация оснований — это механизм, используемый для обнаружения и удаления определенных типов поврежденных азотистых оснований. Ключевую роль в нем играет группа ферментов, называемых гликозилазами. Каждая гликозилаза обнаруживает и удаляет определенный вид поврежденных оснований.
Например, в процессе реакции дезаминирования цитозин может превратиться в урацил — основание, обычно встречающееся только в РНК. Во время репликации ДНК урацил будет соединяться с аденином, а не с гуанином (в отличие от цитозина), поэтому такое превращение может привести к возникновению мутацииstart superscript, 5, end superscript.
Для предотвращения подобных изменений гликозилаза, являющаяся частью сигнального пути эксцизионной репарации, обнаруживает и удаляет дезаминированные цитозины. После того, как основание было удалено, удаляется и оставшаяся часть нуклеотида, а другие ферменты заполняют пробелstart superscript, 6, end superscript.
Эксцизионная репарация нуклеотидов
Эксцизионная репарация нуклеотидов — это еще один способ удаления и замены поврежденных оснований. В результате нее обнаруживаются и корректируются повреждения, которые искажают форму двойной спирали ДНК. Например, азотистые основания могут измениться, присоединив к себе громоздкие группы атомов, в частности, в результате воздействия химических веществ, содержащихся в сигаретном дымеstart superscript, 7, end superscript.
Эксцизионная репарация нуклеотидов также используется для устранения повреждений, вызванных ультрафиолетовым излучением, например, при получении солнечного ожога. Под воздействием УФ-излучения цитозин и тимин могут вступать в реакцию с соседними основаниями, которые также являются цитозином или тимином, образуя при этом связи, изменяющие форму двойной спирали и вызывающие ошибки в процессе репликации ДНК. Наиболее распространенный тип таких связей — тиминовый димер — он состоит из двух тиминовых оснований, вступающих в реакцию друг с другом и образующих химическую связьstart superscript, 8, end superscript.
При эксцизионной репарации нуклеотидов поврежденные нуклеотиды удаляются вместе с соседними нуклеотидами. В этом процессе хеликаза (фермент, раскручивающий ДНК) раскрывает ДНК, образуя пузырь, а ферменты, разрезающие ДНК, отсекают поврежденную часть пузыря. Полимераза заполняет пробел, а лигаза сшивает разрыв в цепиstart superscript, 9, end superscript.
Репарация двухцепочечных разрывов
Некоторые факторы окружающей среды, например, радиация, могут вызывать разрывы обеих цепочек ДНК (разделение хромосомы на две части). Такие повреждения ДНК, если верить комиксам, ведут к появлению супергероев, но могут встречаться и после реальных катастроф, например, Чернобыльской.
Двухцепочечные разрывы опасны, потому что большие сегменты хромосом и сотни содержащихся в них генов могут быть потеряны, если разрыв не будет восстановлен. Существует два способа восстановления двухцепочечных разрывов ДНК: негомологичное соединение концов и гомологичная рекомбинация.
При негомологичном соединении концов два разорванных конца хромосомы просто склеиваются обратно. Этот механизм восстановления является «грубым» и неточным, в результате в месте разрыва, как правило, либо теряются нуклеотиды, либо добавляются лишние, что может привести к мутациям. Но это в любом случае лучше потери целого фрагмента хромосомыstart superscript, 10, end superscript.
При гомологичной рекомбинации для восстановления разрыва используется фрагмент из гомологичной хромосомы, который соответствует поврежденной хромосоме (или из сестринской хроматиды, если ДНК была реплицирована). В этом процессе две хромосомы объединяются, и неповрежденная область гомологичной хромосомы или хроматиды используется в качестве матрицы для замены поврежденной области. Гомологичная рекомбинация работает «чище», точнее, чем негомологичное соединение концов, и обычно не приводит к образованию мутацийstart superscript, 11, end superscript.
Репарация ДНК и заболевания человека
Доказательства важности механизмов репарации получены на основе генетических заболеваний человека. Во многих случаях мутации в генах, которые кодируют белки, участвующие в репарации, связаны с наследственным раком. Например:
-
Наследственный неполипозный колоректальный рак (также называемый синдромом Линча) вызван мутациями в генах, кодирующих белки, которые участвуют в репарации ошибочно спаренных нуклеотидовstart superscript, 12, comma, 13, end superscript. Поскольку такие нуклеотиды не восстанавливаются, у людей, страдающих этим синдромом, мутации накапливаются гораздо быстрее, чем у здоровых. Это может привести к развитию опухолей толстой кишки.
-
Люди с пигментной ксеродермой очень чувствительны к ультрафиолетовому излучению. Это вызвано мутациями в белках, участвующих в эксцизионной репарации нуклеотидов. Когда они не функционируют, димеры тимина и другие виды повреждений, вызванные ультрафиолетовым излучением, перестают восстанавливаться. У людей с пигментной ксеродермой после нескольких минут пребывания на солнце могут возникнуть сильные солнечные ожоги, и около половины из них заболевают раком кожи в возрасте до 10 лет, если только они не избегают солнечных лучейstart superscript, 14, end superscript.
Макеты страниц
Было установлено, что частота ошибок при репликации ДНК Е. coli не превышает 1 на  нуклеотидов. Поскольку хромосома Е. coli содержит приблизительно
нуклеотидов. Поскольку хромосома Е. coli содержит приблизительно  пар оснований, на 10000 клеток, претерпевших один цикл деления, встраивается всего один неправильный нуклеотид.
пар оснований, на 10000 клеток, претерпевших один цикл деления, встраивается всего один неправильный нуклеотид.
Долгое время считалось, что столь высокая степень точности воспроизведения генетической информации целиком определяется точностью уотсон-криковского спаривания между матричной и новообразованной (дочерней) цепями, однако в результате последующего анализа выяснилось, что если бы точность репликации зависела исключительно от правильности спаривания оснований, то частота ошибок была бы значительно выше — приблизительно 1 на 104-105 остатков. Следовательно, чтобы объяснить такую низкую частоту ошибок при репликации in vivo, необходимо предположить участие в процессе репликации еще какого-то одного или нескольких факторов.
Более детальное изучение свойств высокоочищенных ДНК-полимераз позволило получить по крайней мере частичный ответ на вопрос о природе этих факторов. Напомним, что ДНК-полимеразы I и III обладают тремя различными ферментативными активностями. Мы уже видели, как фермент функционирует в качестве полимеразы, а также как он может удалять нуклеотидные остатки с 5-конца фрагмента ДНК. Однако 3-экзонуклеазная активность ДНК-полимераз I и III очень озадачивала исследователей, ибо она означала, что эти ферменты способны «пятиться», отщепляя З-концевые нуклеотиды в направлении, противоположном тому, в котором они действуют как полимеразы. З-экзону-клеазная активность ДНК-полимераз I и III — это средство проверки новосинтезированной цепи ДНК и исправления ошибок, сделанных ферментом при его работе в качестве полимеразы. Если ДНК-полимераза встраивает неправильный нуклеотид, то фермент сам может распознать неспособность этого нуклеотида образовать правильную пару с соответствующим нуклеотидом матрицы (рис. 28-15). В этом случае фермент возвращается назад и отщепляет неправильный нуклеотид с З-конца цепи, после чего полимераза продолжает присоединять правильные нуклеотиды, т.е. возобновляет свое обычное продвижение в направлении 

Рис. 28-15. Исправление ошибок с помощью 3-зкзонуклеазной активности ДНК-полимеразы.
Таким образом, по мере перемещения репликативной вилки вдоль матрицы осуществляется проверка каждого встроенного нуклеотида. Корректирующее действие ДНК-полимеразы очень эффективно; благодаря ему точность репликации повышается как минимум в 104 раз. Суммарная ошибка возникает в результате ошибок, допускаемых ферментом в ходе полимеризации и в процессе исправления их при корректировке; она не превышает одной ошибки на  нуклеотидных остатков.
нуклеотидных остатков.
Очень важно отметить, что процесс репликации протекает со значительно более высокой степенью точности, чем процессы транскрипции и трансляции. Частые ошибки в репликации подвергли бы большому риску сохранность видов и их жизнеспособность.
Ошибки же в транскрипции и трансляции гораздо менее опасны, поскольку они влияют на образование РНК или белка только в одной клетке и не изменяют всю последующую родословную вида. Корректировка с помощью ДНК-полимеразы — это, вероятно, лишь один из путей, обеспечивающих высокую точность репликации. Возможно, исключительно сложная организация репликативного процесса и участие в нем множества белков необходимы для достижения именно этой цели. Интересно, что некоторые эукариотические ДНК-полимеразы не осуществляют корректировку. По-видимому, эукариоты обеспечивают точность и надежность процесса репликации с помощью каких-то других средств.
Удвоение генетического материала перед делением клетки — очень точный процесс. Но мутации в геноме не перестают накапливаться, что приводит как к болезням, так и к появлению нового материала для эволюции. Оказывается, одним из механизмов сохранения мутаций может служить связывание белков — полимераз и транскрипционных факторов — с ДНК. Они создают помеху для «выщепления» ошибочных последовательностей, синтезированных неточной ДНК-полимеразой α.
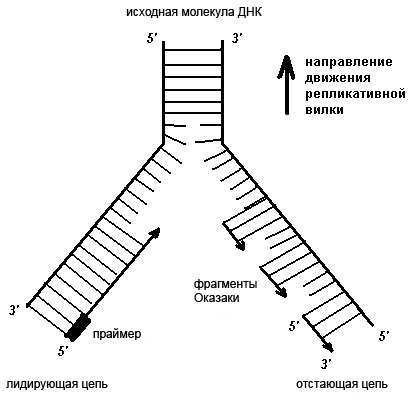
Рисунок 1. Схема репликативной вилки. Новая цепь синтезируется на матрице старой в направлении от 5’- к 3’-концу. При этом одна из цепей может расти непрерывно, а другая — нет (очередное праймирование происходит по мере расплетения ДНК). Рисунок с сайта topreferat.znate.ru.
Перед тем как поделиться, клетке нужно дублировать свой генетический материал. Процесс удвоения молекул ДНК называется репликацией. Репликация осуществляется и регулируется многими белками. Она должна быть максимально точной, чтобы избежать появления мутаций в ДНК. Механизм удвоения генетического материала у прокариот и эукариот полуконсервативный. То есть после репликации одной двуцепочечной молекулы ДНК получаются две, в каждой из которых одна цепь будет старой (материнской), а другая новой (дочерней) [1]. Образовавшиеся молекулы ДНК почти идентичные. Но их минорные отличия могут играть важную роль в судьбе организма [2, 3].
Немного о ДНК-полимеразах
Ключевыми белками репликации являются ДНК-полимеразы — ферменты, читающие цепочку нуклеотидов и использующие ее как матрицу для синтеза дочерней молекулы ДНК. Важно отметить, что ДНК-полимеразы могут присоединять новый нуклеотид только к предыдущему, потому им всегда нужна «затравка» (праймер). Такие затравки во время репликации синтезирует другой фермент — праймаза. Издержки такого разделения труда заключаются в том, что первым компонентом дочерней цепи становятся рибонуклеотиды, то есть короткие цепочки РНК. К ним полимераза уже может присоединить дезоксирибонуклеотиды. Это делает ДНК-полимераза α. Она удлиняет РНК-затравку, синтезируя небольшую цепочку ДНК (примерно 20 нуклеотидов). Так как репликация должна быть очень точной, то неудивительно, что некоторые ДНК-полимеразы умеют исправлять ошибки своей работы. В первую очередь это касается ферментов, синтезирующих длинные участки цепей. Полимераза α такой редактирующей способностью не обладает, поэтому включает новые нуклеотиды, не «оглядываясь» назад и оставляя много ошибок.
Каждая цепь ДНК имеет направление: у нее есть 3’- и 5’-концы. Важно отметить, что новая молекула ДНК может удлиняться только в одну сторону, так как ДНК-полимеразы умеют добавлять нуклеотиды только к 3’-концу. В начале репликации две цепи материнской ДНК расплетаются в определенном месте, и на их матрицах строятся дочерние цепи в двух направлениях. В одну сторону синтез идет непрерывно, а в другую — с помощью коротких фрагментов, как показано на рисунке 1. Такой механизм обеспечивает удлинение дочерних цепей только от 5’-конца к 3’-концу [4, 5]. Цепь, синтез которой идет короткими фрагментами — фрагментами Оказаки, называется отстающей.
У эукариот синтез всего фрагмента Оказаки от уже продленной полимеразой α затравки осуществляет полимераза δ. Она очень точная и умеет исправлять свои ошибки. Получается, что в начале каждого фрагмента Оказаки есть небольшой фрагмент РНК и участок, синтезированный «малограмотной» полимеразой α. Всё это нужно удалить и застроить заново полимеразой δ, так как ни РНК, ни ошибок в дочерней цепи быть не должно (рис. 2). Поле такого редактирования фрагменты Оказаки сшиваются ДНК-лигазой, образуя непрерывную цепь [6, 7]. Но, несмотря на исправление ошибок, некоторые из них все-таки остаются, и возникают мутации.
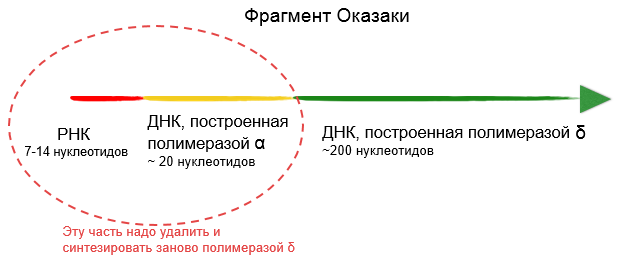
Рисунок 2. Структура фрагмента Оказаки. В начале фрагмента есть часть, которая особенно нуждается в редактировании (ограничена пунктиром). В клетке для этого имеются специальные механизмы. Но если с ДНК быстро свяжется белок, то ошибки в 5’-концевых участках фрагментов Оказаки могут остаться неисправленными.
Возможный механизм сохранения мутаций после репликации
Показано, что мутации в геноме происходят неравномерно [8]. Их распределение зависит от многих факторов, в том числе и от деятельности ДНК-связывающих белков. Авторы недавнего исследования сопоставили распределение мутаций в геноме и участков «стыка» фрагментов Оказаки (рис. 3а). Получилось, что на центр нуклеосомной ДНК приходится как пик мутаций, так и максимальное количество концов фрагментов Оказаки [9].
Такая корреляция была обнаружена и в зонах связывания транскрипционных факторов (рис. 3b). Правда, непосредственно специфические сайты посадки белков мутируют редко. Это, видимо, связано с давлением отбора: если сайт связывания транскрипционного фактора будет поврежден, то может сбиться регуляция важных процессов, что в свою очередь с высокой вероятностью приведет к гибели клетки.
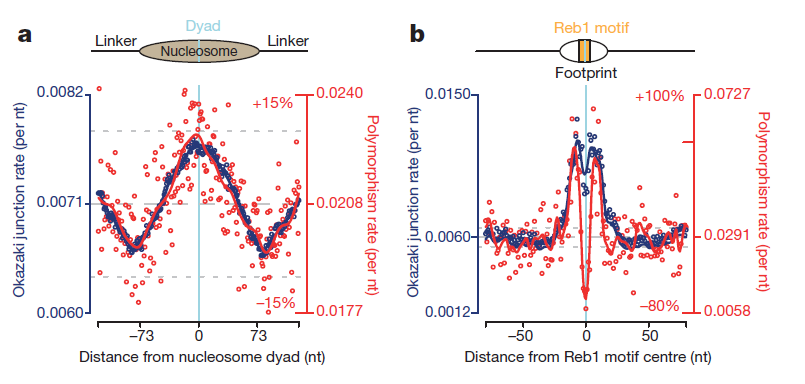
Рисунок 3. Корреляция нуклеотидного полиморфизма (числа мутаций) и количества стыков фрагментов Оказаки в местах посадки белков (нуклеосомных — слева, транскрипционных регуляторов — справа) на ДНК дрожжей S. cerevisiae. Рисунок из [9].
Синтез фрагментов Оказаки имеет направление. Если мутации связаны с этим процессом, то их распределение тоже должно иметь направление. Выявили, что количество мутаций резко возрастает сразу после окончания фрагмента Оказаки. Особенно в местах связывания белков, служащих барьером для полимеразы δ [10].
Авторы показали, что совпадение мест стыка фрагментов Оказаки и повышенного уровня мутаций является следствием именно того, что в этих участках с ДНК связываются белки. Последовательности ДНК в таких регионах всегда разные, поэтому не имеют прямого отношения к наблюдаемой закономерности.
Как было сказано выше, фрагменты Оказаки синтезируются полимеразами α и δ. Ранее полагали, что в ходе этого процесса большинство нуклеотидов (если не все), синтезированных полимеразой α, удаляются [11, 12]. Это было бы полезно, потому что у полимеразы α нет возможности исправления ошибок. Но авторы исследования предполагают, что «следы» полимеразы α будут оставаться, если с ДНК быстро свяжется белок (рис. 4).
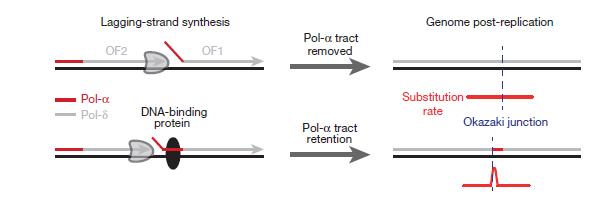
Рисунок 4. Модель закрепления ошибок репликации из-за белка, связанного с началом фрагмента Оказаки. Рисунок из [9].
В подтверждение этой гипотезы ученые показали, что ДНК, синтезированная полимеразой α, остается в геноме и после завершения репликации, а не исключается целиком. Мало того, она составляет 1,5% генома.
Интересно, что найденные закономерности характерны не только для дрожжей, но и для человека. Часто вокруг типичных сайтов связывания белков в ДНК накапливаются мутации. Авторы исследования связывают это именно с активностью полимеразы α.
- Meselson M., Stahl F.W. (1958). The replication of DNA in Esherichia coli. Proc. Natl. Acad. Sci. USA. 44 (7), 671–682;
- Генная терапия против рака;
- Союз голубоглазых;
- Johnston L.H., Nasmyth K.A. (1978). Saccharomyces cerevisiae cell cycle mutant cdc9 is defective in DNA ligase. Nature. 274, 891–893;
- Okazaki R., Okazaki T., Sakabe K., Sugimoto K., Sugino A. (1968). Mechanism of DNA chain growth. I. Possible discontinuity and unusual secondary structure of newly synthesized chains. Proc. Natl. Acad. Sci. USA. 59, 598–605;
- Balakrishnan L. and Bambara R.A. (2013). Okazaki fragment metabolism. Cold Spring Harb. Perspect. Biol. 5 (2), doi: 10.1101/cshperspect.a010173;
- Zheng L. and Shen B. (2011). Okazaki fragment maturation: nucleases take centre stage. J. Mol. Cell Biol. 3, 23–30;
- Wolfe K.H., Sharp P.M., Li W.H. (1989). Mutation rates differ among regions of the mammalian genome. Nature. 337, 283–285;
- Reijns M.A.M., Kemp H., Ding J., de Proce S.M., Jackson A.P., Taylor M.S. (2015). Lagging-strand replication shapes the mutational landscape of the genome. Nature. 518, 502–506;
- Smith D.J. and Whitehouse I. (2012). Intrinsic coupling of lagging-strand synthesis to chromatinassembly. Nature. 483, 434–438;
- Perera R.L., Torella R., Klinge S., Kilkenny M.L., Maman J.D., Pellegrini L. (2013). Mechanism for priming DNA synthesis by yeast DNA Polymerase alpha. eLife 2013;2:e00482;
- Walsh E. and Eckert K.A. Eukaryotic replicative DNA Polymerases. In: Murakami K. and Trakselis M.A. (Eds.), Nucleic Acid Polymerases. Springer-Verlag Berlin Heidelberg, 2014. 30, 17–41. ISBN 978-3-642-39795-0..
Исправление ошибок. ДНК-полимеразы могут выявлять и исправлять ошибки, тогда как РНК-полимеразы такой способностью, по-видимому, не обладают. Поскольку ошибка даже в одном основании как при репликации, так и при транскрипции может привести к ошибке в синтезе белка, можете ли вы дать биологическое объяснение этому поразительному различию [c.925]
З-Ю п. н. Оказывается, у всех организмов точность работы репликативной машины (включающей не только ДНК-полимеразы, но и другие белки см. ниже) как раз такова, чтобы обеспечить безошибочное воспроизведение всего генома или допустить лишь малое число ошибок. Так, у бактерий ошибки синтеза ДНК происходят не чаще чем один раз на много миллионов нуклеотидов. Молекулярные взаимодействия, на которых основаны ферментативные реакции, в частности синтез ДНК, не могут быть абсолютно надежными, кроме того, точность процесса связана с его скоростью. Для того чтобы обеспечить высокую точность наряду с высокой скоростью репликации, природе пришлось прибегнуть к специальным механизмам, один из которых — механизм коррекции. [c.47]
Смещение в комплементарности пар, или ошибка репликации может происходить по другой схеме [c.219]
Нормальное размножение клеток требует высокой точности копирования ДНК-матрицы. Генетический материал живых организмов имеет огромные размеры. Даже у бактерий ДНК-полимераза должна практически безошибочно скопировать молекулу ДНК длиной около 3-10 п. н. Оказывается, у всех организмов точность работы репликативной машины (включаюш.ей не только ДНК-полимеразы, но и другие белки см. ниже) как раз такова, чтобы обеспечить безошибочное воспроизведение всего генома или допустить лишь малое число ошибок. Так, у бактерий ошибки синтеза ДНК происходят не чаще чем один раз на много миллионов нуклеотидов. Молекулярные взаимодействия, на которых основаны ферментативные реакции, в частности синтез ДНК, не могут быть абсолютно надежными, кроме того, точность процесса связана с его скоростью. Для того чтобы обеспечить высокую точность наряду с высокой скоростью репликации, природе пришлось прибегнуть к специальным механизмам, один из которых — механизм коррекции. [c.47]
Очень важно отметить, что процесс репликации протекает со значительно более высокой степенью точности, чем процессы транскрипции и трансляции. Частые ошибки в репликации подвергли бы большому риску сохранность видов [c.908]
ДНК-полимеразы проверяют комплементарность каждого нуклеотида матрице дважды один раз перед включением его в состав растущей цепи и второй раз перед тем, как включить следующий нуклеотид. Очередная фосфодиэфирная связь образуется лишь в том случае, если последний (З -концевой) нуклеотид затравки комплементарен матрице. Если же на предыдущей стадии полимеризации произошла ошибка (например, из-за того, что нуклеотид в момент полимеризации находился в необычной таутомерной форме), то репликация останавливается до тех пор, пока неправильный нуклеотид не будет удален. Некоторые ДНК-полимеразы обладают не только полимеризующей, но и 3 -экзонуклеазной активностью, «Которая отщепляет не спаренный с матрицей нуклеотид затравки. После чего полимеризация восстанавливается, от механизм, коррекция, заметно увеличивает точность работы ДНК-полимераз. Мутации, нарушающие З -экзонуклеазную активность ДНК-полимеразы, существенно повышают частоту возникновения прочих мутаций. Напротив, мутации, приводящие к усилению экзонуклеазной актив- ности относительно полимеризующей, снижают темп мутирования Генетического материала. [c.47]
Спонтанные генные мутации определяются ошибками при репликации ДНК, возникающими вследствие теплового движе-иия атомов и молекул. Очевидно, что ошибки транскрипции и трансляции не наследуются. [c.283]
Процесс транскрипции находится в клетке под строгим контролем, поэтому имеет место как неодинаковое транскрибирование во времени разных участков ДНК (генов), так и неодинаковая скорость, с которой гены могут транскрибироваться. В результате количество молекул иРНК в клетке, комплементарных разным генам, сильно различается. Хотя в целом механизмы синтеза ДНК и РНК сходны, процесс транскрипции не обладает той степенью точности, которая характерна для репликации ДНК. Однако поскольку иРНК не способна к самовоспроизведению, возникающие при ее синтезе ошибки в последующих клеточных генерациях не воспроизводятся и, следовательно, не могут наследоваться. [c.142]
Если ошибка синтеза не устраняется системами репарации, то неизбежна деформация дуплекса и искажение генетической программы. Такие сохраняющиеся при репликации изменения ДНК носят название мутации. Они могут быть спонтанными и индуцированными. Частота спонтанных мутаций невелика и составляет всего 10 —10 на клетку. В основном имеют место мутации, обусловленные действием внешних факторов физических (радиация), биологических (вирусы) и чужеродных химических веществ на генетический аппарат клеток. Наиболее многочисленными и опасными являются мутагены окружающей среды. Загрязнение воды и воздуха различными химическими отходами промышленных предприятий, химическими средствами защиты растений отрицательно сказывается на генетической программе всех живых организмов. В последние годы установлено, что ряд пищевых красителей, стабилизаторов и вкусовых добавок обладает выраженной мутагенной активностью, что привело к значительному ужесточению требований, связанных с применением химических веществ в пищевой промышленности. Многие лекарственные вещества также воздействуют на генетический аппарат клеток и должны подвергаться специальным генетическим испытаниям. [c.455]
Химическое изменение оснований. Некоторые мутагенные вещества действуют путем химического изменения содержащихся в ДНК оснований, что приводит к ошибкам репликации. Вполне понятное изменение вызывает нитрит. Азотистая кислота дезаминирует аденин, гуанин или цитозин без разрыва или каких-либо других изменений полинуклеотидной цепи. В результате замещения аминогруппы гидроксильной группой аденин превращается в гипоксантин и спаривается с цитозином вместо тимина, что приводит к мутации АТ СС. Если цитозин дезаминируется в урацил, то он спаривается с аденином вместо гуанина, и это ведет к мутации СС -АТ. Будучи превращен в ксантин, гуанин по-прежнему спаривается с цитозином, т. е. дезаминирование С не вызывает мутации. Гидроксиламин вступает в реакцию главным образом с цитозином и изменяет его так, что тот спаривается с аденином значит, он тоже вызывает мутации СС ТА. [c.444]
Из уровня спонтанных мутаций у бактерий в расчете на одно поколение рассчитано, что вероятность одной репликационной ошибки при синтезе ДНК составляет порядка 10 . Эту величину можно рассматривать как отношение скоростей реакций правильной репликации [c.194]
ВЫВОД, ЧТО, по-видимому, код действительно является триплет-ным, причем кодирование начинается от определенной точки нуклеиновой кислоты. При этом большая часть трехбуквенных комбинаций соответствует определенным аминокислотам и лишь небольшая часть триплетов относится к бессмысленным. Число триплетов равно 4-4-4 = 64, т. е. значительно больше числа аминокислот. Некоторые из них, по-видимому, кодируют одну и ту же аминокислоту, т. е. код является вырожденным. Этот вывод согласуется с обнаружением в настоящее время двух и более типов растворимых РНК, специфичных к одной и той же аминокислоте. Вырожденность генетического кода может способствовать выживанию организма. Действительно, в случае невырожденного кода ошибка при репликации ДНК или при транскрипции должна скорее приводить к появлению бессмысленного триплета, чем в случае вырожденного кода. Следовательно, при невырожденном коде ошибки чаще вызывали бы прекращение синтеза соответствующего белка или образование незаконченных белковых цепей. Напротив, в случае вырожденного кода ошибки должны чаще приводить просто к замене одной аминокислоты на другую, что, как правило, не имеет серьезных последствий. [c.376]
Как в прокариотических, так и в эукариотических клетках содержатся ферментные системы, способные исправлять ошибки репликации и различные формы повреждения ДНК, вызываемые гидро- [c.990]
Г. Е. Фрадкин. После обработки фаговой популяции гидроксиламино.м последний при помощи диализа удалялся из вирусной суспензии. Следовательно, во время облучения гидроксиламин в среде отсутствовал. Предварительная модификация цитозиновых остатков в ДНК фага лямбда, вызываемая гидроксиламином (предположительно образование 4—5-дигидро-4-гидро-ксиламиноцитозина), действительно повышает радиочувствительность фаговой популяции в условиях преобладания непрямого эффекта излучения. Мы полагаем, что механизм повышения радиочувствительности сводится к нарушению специфического процесса комплементарного спаривания азотистых оснований во время репликации фаговой ДНК внутри клетки. В последних рабо тах Брауна, Филипса с соавторами химическими методами установлено, что цитозин, предварительно обработанный гидроксиламином, спаривается не с гуанином, а с аденином. Вследствие этого во вновь образованной ДНК происходят единичные замены гуанина на аденин. До тех пор, пока эти замены не выходят за пределы связанных серий однозначных кодонов, они не сказываются на информационных свойствах ДНК фага. Однако эти единичные замены понижают эффективность механизма, исправляющего ошибки включения, за счет уменьшения резерва однозначны кодонов или, иными словами, за счет уменьшения степени вырожденности структурного кода. Мы не видим большой сложности в этом объяснении, к которому мы сознательно прибегли для освещения возмол<ных молекулярных механизмов, лежащих в основе скрытых повреждений, связанных с тонкими сдвигами в величинах водородных сил в химически модифицированных азотистых основаниях. Как известно, сенсибилизация может обусловливаться уменьшением степени прочности первичной структуры ДНК вследствие лабилизации эфирно-фосфатных связей. Однако при использовании в качестве модифицирующего агента гидроксиламина этот второй механизм отсутствует, так как химическими исслг- [c.173]
Включение или утрата отдельных пар оснований. Профлавин и другие акридиновые красители действуют по-иному. Вероятно, молекула акридина внедряется между соседними основаниями цепи ДНК и увеличивает расстояние между ними (интеркаляция). Такое пространственное изменение при репликации ДНК может вызывать ошибки двух типов- [c.444]
НО редко, он может образовать пару не с аденином, а с гуанином. Это в свою очередь приведет к ошибке при включении или при репликации, заключающейся в замене пары А — Т на Г — Ц и наоборот. [c.218]
В 1959 г. Д. Пратт сумел показать, что большинство, если не все бромурациловые ревертанты г+, образуемые мутантами гП (которые были индуцированы аналогами оснований), возникают в виде гетерозигот гП/г» , которые позднее расщепляются на гомозиготные ревертанты г» «. Чтобы продемонстрировать это, к бактериям, зараженным мутантным фагом Т4гП, непосредственно перед окончанием скрытого периода внутриклеточного развития фага добавляли бромурацил и первые инфекционные частицы, появившиеся в клетках непосредственно после окончания скрытого периода, высвобождали путем искусственного лизиса клеток. Такая методика постановки опыта гарантировала, что все ревертанты / +, возникшие и извлеченные из фонда предшественников фаговой ДНК во время короткого воздействия мутагена, образовались исключительно в самом последнем цикле репликации. Ошибка копирования, восстановившая у них в соответствующем участке ДНК генетическую информацию дикого типа г+, произошла настолько поздно, что больше и и одного цикла репликации произойти уже не могло (а это значит, что не могло произойти и расщепления на гомозиготные мутантные структуры). Такого рода опыты показали, что свыше 80% всех ревертантов г, возникших в результате кратковременного контакта с бромурацилом, действительно представляет собой мутационные гетерозиготы, несущие как исходный аллель г, так и ревертировавщий к дикому типу аллель г» . Следовательно, в полном соответствии с механизмом Уотсона и Крика и вопреки механизмам, предусматривающим консервативное распределе- [c.325]
Нетрудно видеть, что в тонком механизме репликации и синтеза белков произвол в расположении частиц сведен к минимуму. Этот матричный процесс является низкоэнтропийным. Ошибки в размещении аминокислот в пептидных цепочках составляют по приблизительной оценке 1 на 10 . В то же время, если бы синтез белков происходил на примитивной матрице, на которой концентрация тех или иных компонентов и их относительное расположение в значительной мере определялись бы случайностями окружающей обстановки, нельзя было бы ожидать воспроизводимости синтеза того или иного белка и, в частности, того белка, от структуры ко- [c.393]
Репликаза фага Q способна in vitro синтезировать цепи, полностью комплементарные как плюс-, так и минус-молекулам вирусной РНК. Система, однако, специфична для вирусной РНК и не может копировать никаких других полинуклеотидов. Возможно, что для инициации процесса репликации нужно, чтобы на З -конце имелись определенные последовательности. В пробирке репликация протекает с ошибками, такими, в частности, как преждевременная терминация цепи и неправильное спаривание оснований. В результате происходит образование мутантных форм РНК, что дает возможность получать молекулы РНК, размеры которой будут значительно меньше, чем у вирусной РНК, и которые будут при этом легко реплицироваться репликазной системой фага Q . Была установлена нуклеотидная последовательность одного из таких фрагментов, включающего всего лишь 114 нуклеотидов . [c.245]
Разные аллели одного и того же Г. возникают благодаря мутациям-илслецуемьш изменениям в структуре исходного Г. В норме Г. чрезвычайно стабилен и при удвоении хромосом во время репликации ДНК воспроизводится совершенно точно вероятность ошибки не превышает 10″ . Мутации происходят редко и обычно влекут за собой неблагоприятные последствия для организма, т. к. нарушается его способность синтезировать нормальный белок. Однако в целом это явление играет положит, роль накопление редких полезных мутаций создает основу генетич. изменчивости, необходимой для эволюции. [c.517]
Особый класс М. составляют соед., представляющие собой аналоги оснований ДНК-5-галогенурацилы, 2-амино-и 6-метиламинопурины н др. Галогенурацилы включаются в ДНК при матричном синтезе вместо тимина, 2-амино-пурин-вместо аденина. Вследствие различий в положении кетоенольного равновесия у тимина и галогенурацилов (при включении последних в ДНК) увеличивается частота ошибочных спариваний оснований и возникают ошибки при репликации. [c.152]
Равновесие между созидательными возможностями выбора среди специфических оснований в ДНК (созидательные мутации) и точностью синтеза белков (поддерживающих жизнь организма) является основой эволюции. Ферменты, которые заряжают тРНК специфической аминокислотой, обладают очень низкой вероятностью ошибки, порядка 1 Ю» для гомологичных аминокислот. При репликации точность даже выше, и величина ошибки редко превышает 1 на 10. [c.212]
К настоящему времени у эукариот, как и у бактерий (см. ранее), открыто несколько ДНК-полимераз. В репликации ДНК эукариот участвуют два главных типа полимераз — а и б. Показано, что ДНК-полимераза а состоит из 4 субъединиц и является идентичной по структуре и свойствам во всех клетках млекопитающих, причем одна из субъединиц оказалась наделенной праймазной активностью. Самая крупная субъединица ДНК-полимеразы а (мол. масса 180000) катализирует реакцию полимеризации, преимущественно синтез отстающей цепи ДНК, являясь составной частью праймасомы. ДНК-полимераза б состоит из 2 субъединиц и преимущественно катализирует синтез ведущей цепи ДНК (см. далее). Открыта также ДНК-полимераза г, которая в ряде случаев заменяет б-фермент, в частности при репарации ДНК (исправление нарушений ДНК, вызванных ошибками репликации или повреждающими агентами). Следует отметить, что в эукариотических клетках открыты два белковых фактора репликации, обозначаемых RFA и RF . Фактор репликации А выполняет функцию белка—связывание одноцепочечной ДНК (наподобие белковых факторов связывания разъединенных цепей ДНК при [c.480]
Этап HI — терминация синтеза ДНК —наступает, скорее всего, когда исчерпана ДПК-матрица и трансферазные реакции прекращаются. Точность репликации ДНК чрезвычайно высока, возможна одна ошибка на 10 трансферазных реакций, однако подобная ошибка обычно легко исправляется за счет процессов репарации. [c.486]
Такие системы, достигшие определенного уровня сложности, наталкиваются на границу генетически переносимого количества информации. Оно составляет около 10 бит. Оптимальное значение ошибки при репликации нуклеиновой кислоты — 10 . Это — системно-обусловленная граница. Ее преодоление было достигнуто в нрироде созданием пола и геыегической рекомоина- [c.549]
Приближенная модель репликации ДНКизображена на рис. 2.11. Из приведенной схемы видно, что репликация точно воспроизводит прежнюю (исходную) структуру ДНК. Но если произошла ошибка в процессе копирования (мутация), то она будет с предельной точностью копироваться при последующих репликациях изменившейся ДНК. Показано, что участки ДНК, содержащие скопления нуклеотидов, обладают повышенной склонностью к спонтанным мутациям [22]. [c.94]
По происхождению мутации делятся на спонтанные (неконтролируемые) и индуцированные (контролируемые). Первые возникают в результате неконтролируемого влияния каких-то естественных факторов (радиация, температура и т. д.). Направленное использование мутагенов приводит к возникновению индуцированных мутаций. Многими экспериментами четко показано, что мутации возникают независимо от условий среды обитания, т. е. не направленно. Мутации возникают в основном как ошибки репликации ДНК. Выделяют следующие типы мутаций перестройка хромосом, перестройка генома клетки грибов и водорослей (полиплоидия, гаплоидия, гетероплоидия), внутригенные изменения (прямые мутации, реверсии, обратные мутации). [c.102]
Ошибка в одном основании при репликации ДНК, если она не исправлена, приведет к тому, что одна из двух дочерних клеток, а также все ее потомки будут содержать измененную хромосому. Ошибка в одном основании, совершенная РНК-полимеразой, повлечет за собой синтез некоторого количества неправильных копий одного белка. При этом, поскольку пул мРНК в клетке быстро обновляется, большинство молекул этого белка будет нормальным. Потомство такой клетки тоже будет нормальным. [c.1004]
Анализ приведенных выше результатов дает возможность написать для преобладающих таутомерных форм оснований нуклеиновых кислот формулы, изображенные на фиг. 55. Минорные таутомерные формы, возможно, играют существенную роль в возникновении спонтанных мутаций, поскольку спаривание несоответствующих оснований (см. гл. ХУП1) должно привести к ошибке при включении оснований и при последующей репликации цепи. Можно показать, что если скорость включения основания в цепь нуклеиновой кислоты меньше скорости перехода минорного таутомера в доминирующую форму, то скорость спонтанных мутаций, обусловленных данным основанием, приблизительно равна константе равновесия между минорным и доминирующим таутомерами. К сожалению, для азо- [c.308]
Если ДНК представляет собой генетический материал, то возникает весьма важный вопрос каким образом ДНК реплицируется столь точно, что при передаче генетических признаков очень редко возникают ошибки Так как количество ДНК, приходящееся на гаплоидный набор хромосом, есть величина постоянная, делящаяся клетка должна синтезировать ДНК- Для того чтобы наследственная информация, содерлсащаяс в ДНК, была передана без ошибок, вновь синтезированная ДНК должна представлять собой точную копию исходной. На фиг. 61 изображены схемы двух предполагаемых типов репликации ДНК консервативного и полуконсервативного. [c.327]
Задание 189. Напишите программу для моделирования самоорганизации ДНК в качестве примера самоорганизуюшихся систем. Используйте для этого следующую простую модель. Пусть имеется 100 молекул ДНК, состоящих из 12 нуклеотидов четырех видов (их обозначим буквами А, Т, С и G). Последовательность нуклеотидов в этих 100 молекулах ДНК случайная. Назовем одну из последовательностей идеальной она имеет некоторые преимущества перед остальными. Из 100 молекул ДНК в результате репликации получается еще 100 молекул. Однако при репликации встречаются ошибки (мутации), например в количестве 1%. Теперь из 200 молекул 100 погибает. При этом имеет значение преимущество, которым обладают молекулы с последовательностью нуклеотидов, похожей на идеальную . (Например, при каждом совпадении нуклеотида и его положения в цепи с идеальной последовательностью вероятность гибели уменьшается в два раза.) Процессы репликации и гибели протекают очень быстро. В конце концов все молекулы ДНК должны получить идеальную последовательность нуклеотидов, хотя вероятность ее образования в результате случайного процесса составляет 1 16777216. Что будет, если мутации будут возникать чаще или реже [c.330]
Нетрудно видеть, что в тонком механизме репликации и синтеза белков произвол в расположении частиц сведен к минимуму. Этот матричный процесс является низкоэнтропийным. Ошибки в размещении аминокислот в пептидных цепочках составляют по приблизительной оценке 1 на 10 . В то же время, если бы синтез белков происходил на примитивной матрице, на которой концентрация тех или иных компонентов и их относительное расположение в значительной мере определялись бы случайностями окружающей обстановки, нельзя было бы ожидать воспроизводимости синтеза того или иного белка и, в частности, того белка, от структуры которого зависят жизненно важные свойства системы. Здесь кодирование матричного синтеза обусловлено целым рядом низших кодов кодом, отвечающим соответствию т-РНК и аминокислоты кодом, соответствующим отношению между т-РНК, рибосомой и м-РНК кодом ферментов, производящих замыкание пептидных связей, и т. д. Это — кодированный перенос массы, обусловливающий возникновение структуры, обладающей исключительными свойствами их исключительность состоит в том, что они необходимы для стабилизации синтеза этой же структуры на уровне всех не только низших, но и многих высших кодов, которые возникнут, когда белки сложатся в клетки, клетки в органы, а органы в организм. [c.205]
| DNA-directed DNA polymerase | |||||||
|---|---|---|---|---|---|---|---|

3D structure of the DNA-binding helix-turn-helix motifs in human DNA polymerase beta (based on PDB file 7ICG) |
|||||||
| Identifiers | |||||||
| EC no. | 2.7.7.7 | ||||||
| CAS no. | 9012-90-2 | ||||||
| Databases | |||||||
| IntEnz | IntEnz view | ||||||
| BRENDA | BRENDA entry | ||||||
| ExPASy | NiceZyme view | ||||||
| KEGG | KEGG entry | ||||||
| MetaCyc | metabolic pathway | ||||||
| PRIAM | profile | ||||||
| PDB structures | RCSB PDB PDBe PDBsum | ||||||
| Gene Ontology | AmiGO / QuickGO | ||||||
|
A DNA polymerase is a member of a family of enzymes that catalyze the synthesis of DNA molecules from nucleoside triphosphates, the molecular precursors of DNA. These enzymes are essential for DNA replication and usually work in groups to create two identical DNA duplexes from a single original DNA duplex. During this process, DNA polymerase «reads» the existing DNA strands to create two new strands that match the existing ones.[1][2][3][4][5][6]
These enzymes catalyze the chemical reaction
- deoxynucleoside triphosphate + DNAn ⇌ pyrophosphate + DNAn+1.
DNA polymerase adds nucleotides to the three prime (3′)-end of a DNA strand, one nucleotide at a time. Every time a cell divides, DNA polymerases are required to duplicate the cell’s DNA, so that a copy of the original DNA molecule can be passed to each daughter cell. In this way, genetic information is passed down from generation to generation.
Before replication can take place, an enzyme called helicase unwinds the DNA molecule from its tightly woven form, in the process breaking the hydrogen bonds between the nucleotide bases. This opens up or «unzips» the double-stranded DNA to give two single strands of DNA that can be used as templates for replication in the above reaction.
History[edit]
In 1956, Arthur Kornberg and colleagues discovered DNA polymerase I (Pol I), in Escherichia coli. They described the DNA replication process by which DNA polymerase copies the base sequence of a template DNA strand. Kornberg was later awarded the Nobel Prize in Physiology or Medicine in 1959 for this work.[7] DNA polymerase II was discovered by Thomas Kornberg (the son of Arthur Kornberg) and Malcolm E. Gefter in 1970 while further elucidating the role of Pol I in E. coli DNA replication.[8] Three more DNA polymerases have been found in E. coli, including DNA polymerase III (discovered in the 1970s) and DNA polymerases IV and V (discovered in 1999).[9]
Function[edit]

DNA polymerase moves along the old strand in the 3’–5′ direction, creating a new strand having a 5’–3′ direction.

DNA polymerase with proofreading ability
The main function of DNA polymerase is to synthesize DNA from deoxyribonucleotides, the building blocks of DNA. The DNA copies are created by the pairing of nucleotides to bases present on each strand of the original DNA molecule. This pairing always occurs in specific combinations, with cytosine along with guanine, and thymine along with adenine, forming two separate pairs, respectively. By contrast, RNA polymerases synthesize RNA from ribonucleotides from either RNA or DNA.
When synthesizing new DNA, DNA polymerase can add free nucleotides only to the 3′ end of the newly forming strand. This results in elongation of the newly forming strand in a 5’–3′ direction.
It is important to note that the directionality of the newly forming strand (the daughter strand) is opposite to the direction in which DNA polymerase moves along the template strand. Since DNA polymerase requires a free 3′ OH group for initiation of synthesis, it can synthesize in only one direction by extending the 3′ end of the preexisting nucleotide chain. Hence, DNA polymerase moves along the template strand in a 3’–5′ direction, and the daughter strand is formed in a 5’–3′ direction. This difference enables the resultant double-strand DNA formed to be composed of two DNA strands that are antiparallel to each other.
The function of DNA polymerase is not quite perfect, with the enzyme making about one mistake for every billion base pairs copied. Error correction is a property of some, but not all DNA polymerases. This process corrects mistakes in newly synthesized DNA. When an incorrect base pair is recognized, DNA polymerase moves backwards by one base pair of DNA. The 3’–5′ exonuclease activity of the enzyme allows the incorrect base pair to be excised (this activity is known as proofreading). Following base excision, the polymerase can re-insert the correct base and replication can continue forwards. This preserves the integrity of the original DNA strand that is passed onto the daughter cells.
Fidelity is very important in DNA replication. Mismatches in DNA base pairing can potentially result in dysfunctional proteins and could lead to cancer. Many DNA polymerases contain an exonuclease domain, which acts in detecting base pair mismatches and further performs in the removal of the incorrect nucleotide to be replaced by the correct one.[10] The shape and the interactions accommodating the Watson and Crick base pair are what primarily contribute to the detection or error. Hydrogen bonds play a key role in base pair binding and interaction. The loss of an interaction, which occurs at a mismatch, is said to trigger a shift in the balance, for the binding of the template-primer, from the polymerase, to the exonuclease domain. In addition, an incorporation of a wrong nucleotide causes a retard in DNA polymerization. This delay gives time for the DNA to be switched from the polymerase site to the exonuclease site. Different conformational changes and loss of interaction occur at different mismatches. In a purine:pyrimidine mismatch there is a displacement of the pyrimidine towards the major groove and the purine towards the minor groove. Relative to the shape of DNA polymerase’s binding pocket, steric clashes occur between the purine and residues in the minor groove, and important van der Waals and electrostatic interactions are lost by the pyrimidine.[11] Pyrimidine:pyrimidine and purine:purine mismatches present less notable changes since the bases are displaced towards the major groove, and less steric hindrance is experienced. However, although the different mismatches result in different steric properties, DNA polymerase is still able to detect and differentiate them so uniformly and maintain fidelity in DNA replication.[12] DNA polymerization is also critical for many mutagenesis processes and is widely employed in biotechnologies.
Structure[edit]
The known DNA polymerases have highly conserved structure, which means that their overall catalytic subunits vary very little from species to species, independent of their domain structures. Conserved structures usually indicate important, irreplaceable functions of the cell, the maintenance of which provides evolutionary advantages. The shape can be described as resembling a right hand with thumb, finger, and palm domains. The palm domain appears to function in catalyzing the transfer of phosphoryl groups in the phosphoryl transfer reaction. DNA is bound to the palm when the enzyme is active. This reaction is believed to be catalyzed by a two-metal-ion mechanism. The finger domain functions to bind the nucleoside triphosphates with the template base. The thumb domain plays a potential role in the processivity, translocation, and positioning of the DNA.[13]
Processivity[edit]
DNA polymerase’s rapid catalysis is due to its processive nature. Processivity is a characteristic of enzymes that function on polymeric substrates. In the case of DNA polymerase, the degree of processivity refers to the average number of nucleotides added each time the enzyme binds a template. The average DNA polymerase requires about one second locating and binding a primer/template junction. Once it is bound, a nonprocessive DNA polymerase adds nucleotides at a rate of one nucleotide per second.[14]: 207–208 Processive DNA polymerases, however, add multiple nucleotides per second, drastically increasing the rate of DNA synthesis. The degree of processivity is directly proportional to the rate of DNA synthesis. The rate of DNA synthesis in a living cell was first determined as the rate of phage T4 DNA elongation in phage infected E. coli. During the period of exponential DNA increase at 37 °C, the rate was 749 nucleotides per second.[15]
DNA polymerase’s ability to slide along the DNA template allows increased processivity. There is a dramatic increase in processivity at the replication fork. This increase is facilitated by the DNA polymerase’s association with proteins known as the sliding DNA clamp. The clamps are multiple protein subunits associated in the shape of a ring. Using the hydrolysis of ATP, a class of proteins known as the sliding clamp loading proteins open up the ring structure of the sliding DNA clamps allowing binding to and release from the DNA strand. Protein–protein interaction with the clamp prevents DNA polymerase from diffusing from the DNA template, thereby ensuring that the enzyme binds the same primer/template junction and continues replication.[14]: 207–208 DNA polymerase changes conformation, increasing affinity to the clamp when associated with it and decreasing affinity when it completes the replication of a stretch of DNA to allow release from the clamp.
Variation across species[edit]
| DNA polymerase family A | |||||||
|---|---|---|---|---|---|---|---|

c:o6-methyl-guanine pair in the polymerase-2 basepair position |
|||||||
| Identifiers | |||||||
| Symbol | DNA_pol_A | ||||||
| Pfam | PF00476 | ||||||
| InterPro | IPR001098 | ||||||
| SMART | — | ||||||
| PROSITE | PDOC00412 | ||||||
| SCOP2 | 1dpi / SCOPe / SUPFAM | ||||||
|
| DNA polymerase family B | |||||||
|---|---|---|---|---|---|---|---|

crystal structure of rb69 gp43 in complex with dna containing thymine glycol |
|||||||
| Identifiers | |||||||
| Symbol | DNA_pol_B | ||||||
| Pfam | PF00136 | ||||||
| Pfam clan | CL0194 | ||||||
| InterPro | IPR006134 | ||||||
| PROSITE | PDOC00107 | ||||||
| SCOP2 | 1noy / SCOPe / SUPFAM | ||||||
|
| DNA polymerase type B, organellar and viral | |||||||
|---|---|---|---|---|---|---|---|

phi29 dna polymerase, orthorhombic crystal form, ssdna complex |
|||||||
| Identifiers | |||||||
| Symbol | DNA_pol_B_2 | ||||||
| Pfam | PF03175 | ||||||
| Pfam clan | CL0194 | ||||||
| InterPro | IPR004868 | ||||||
|
Based on sequence homology, DNA polymerases can be further subdivided into seven different families: A, B, C, D, X, Y, and RT.
Some viruses also encode special DNA polymerases, such as Hepatitis B virus DNA polymerase. These may selectively replicate viral DNA through a variety of mechanisms. Retroviruses encode an unusual DNA polymerase called reverse transcriptase, which is an RNA-dependent DNA polymerase (RdDp). It polymerizes DNA from a template of RNA.
| Family[16] | Types of DNA polymerase | Taxa | Examples | Feature |
|---|---|---|---|---|
| A | Replicative and Repair Polymerases | Eukaryotic and Prokaryotic | T7 DNA polymerase, Pol I, Pol γ, θ, and ν | Two exonuclease domains (3′-5′ and 5′-3′) |
| B | Replicative and Repair Polymerases | Eukaryotic and Prokaryotic | Pol II, Pol B, Pol ζ, Pol α, δ, and ε | 3′-5 exonuclease (proofreading); viral ones use protein primer |
| C | Replicative Polymerases | Prokaryotic | Pol III | 3′-5 exonuclease (proofreading) |
| D | Replicative Polymerases | Euryarchaeota | PolD (DP1/DP2 heterodimer)[17] | No «hand» feature, double barrel RNA polymerase-like; 3′-5 exonuclease (proofreading) |
| X | Replicative and Repair Polymerases | Eukaryotic | Pol β, Pol σ, Pol λ, Pol μ, and terminal deoxynucleotidyl transferase | template optional; 5′ phosphatase (only Pol β); weak «hand» feature |
| Y | Replicative and Repair Polymerases | Eukaryotic and Prokaryotic | Pol ι, Pol κ, Pol η,[18] Pol IV, and Pol V | Translesion synthesis[19] |
| RT | Replicative and Repair Polymerases | Viruses, Retroviruses, and Eukaryotic | Telomerase, Hepatitis B virus | RNA-dependent |
Prokaryotic polymerase[edit]
Prokaryotic polymerases exist in two forms: core polymerase and holoenzyme. Core polymerase synthesizes DNA from the DNA template but it cannot initiate the synthesis alone or accurately. Holoenzyme accurately initiates synthesis.
Pol I[edit]
Prokaryotic family A polymerases include the DNA polymerase I (Pol I) enzyme, which is encoded by the polA gene and ubiquitous among prokaryotes. This repair polymerase is involved in excision repair with both 3’–5′ and 5’–3′ exonuclease activity and processing of Okazaki fragments generated during lagging strand synthesis.[20] Pol I is the most abundant polymerase, accounting for >95% of polymerase activity in E. coli; yet cells lacking Pol I have been found suggesting Pol I activity can be replaced by the other four polymerases. Pol I adds ~15-20 nucleotides per second, thus showing poor processivity. Instead, Pol I starts adding nucleotides at the RNA primer:template junction known as the origin of replication (ori). Approximately 400 bp downstream from the origin, the Pol III holoenzyme is assembled and takes over replication at a highly processive speed and nature.[21]
Taq polymerase is a heat-stable enzyme of this family that lacks proofreading ability.[22]
Pol II[edit]
DNA polymerase II is a family B polymerase encoded by the polB gene. Pol II has 3’–5′ exonuclease activity and participates in DNA repair, replication restart to bypass lesions, and its cell presence can jump from ~30-50 copies per cell to ~200–300 during SOS induction. Pol II is also thought to be a backup to Pol III as it can interact with holoenzyme proteins and assume a high level of processivity. The main role of Pol II is thought to be the ability to direct polymerase activity at the replication fork and help stalled Pol III bypass terminal mismatches.[23]
Pfu DNA polymerase is a heat-stable enzyme of this family found in the hyperthermophilic archaeon Pyrococcus furiosus.[24] Detailed classification divides family B in archaea into B1, B2, B3, in which B2 is a group of pseudoenzymes. Pfu belongs to family B3. Others PolBs found in archaea are part of «Casposons», Cas1-dependent transposons.[25] Some viruses (including Φ29 DNA polymerase) and mitochondrial plasmids carry polB as well.[26]
Pol III[edit]
DNA polymerase III holoenzyme is the primary enzyme involved in DNA replication in E. coli and belongs to family C polymerases. It consists of three assemblies: the pol III core, the beta sliding clamp processivity factor, and the clamp-loading complex. The core consists of three subunits: α, the polymerase activity hub, ɛ, exonucleolytic proofreader, and θ, which may act as a stabilizer for ɛ. The beta sliding clamp processivity factor is also present in duplicate, one for each core, to create a clamp that encloses DNA allowing for high processivity.[27] The third assembly is a seven-subunit (τ2γδδ′χψ) clamp loader complex.
The old textbook «trombone model» depicts an elongation complex with two equivalents of the core enzyme at each replication fork (RF), one for each strand, the lagging and leading.[23] However, recent evidence from single-molecule studies indicates an average of three stoichiometric equivalents of core enzyme at each RF for both Pol III and its counterpart in B. subtilis, PolC.[28] In-cell fluorescent microscopy has revealed that leading strand synthesis may not be completely continuous, and Pol III* (i.e., the holoenzyme α, ε, τ, δ and χ subunits without the ß2 sliding clamp) has a high frequency of dissociation from active RFs.[29] In these studies, the replication fork turnover rate was about 10s for Pol III*, 47s for the ß2 sliding clamp, and 15m for the DnaB helicase. This suggests that the DnaB helicase may remain stably associated at RFs and serve as a nucleation point for the competent holoenzyme. In vitro single-molecule studies have shown that Pol III* has a high rate of RF turnover when in excess, but remains stably associated with replication forks when concentration is limiting.[29] Another single-molecule study showed that DnaB helicase activity and strand elongation can proceed with decoupled, stochastic kinetics.[29]
Pol IV[edit]
In E. coli, DNA polymerase IV (Pol IV) is an error-prone DNA polymerase involved in non-targeted mutagenesis.[30] Pol IV is a Family Y polymerase expressed by the dinB gene that is switched on via SOS induction caused by stalled polymerases at the replication fork. During SOS induction, Pol IV production is increased tenfold and one of the functions during this time is to interfere with Pol III holoenzyme processivity. This creates a checkpoint, stops replication, and allows time to repair DNA lesions via the appropriate repair pathway.[31] Another function of Pol IV is to perform translesion synthesis at the stalled replication fork like, for example, bypassing N2-deoxyguanine adducts at a faster rate than transversing undamaged DNA. Cells lacking the dinB gene have a higher rate of mutagenesis caused by DNA damaging agents.[32]
Pol V[edit]
DNA polymerase V (Pol V) is a Y-family DNA polymerase that is involved in SOS response and translesion synthesis DNA repair mechanisms.[33] Transcription of Pol V via the umuDC genes is highly regulated to produce only Pol V when damaged DNA is present in the cell generating an SOS response. Stalled polymerases causes RecA to bind to the ssDNA, which causes the LexA protein to autodigest. LexA then loses its ability to repress the transcription of the umuDC operon. The same RecA-ssDNA nucleoprotein posttranslationally modifies the UmuD protein into UmuD’ protein. UmuD and UmuD’ form a heterodimer that interacts with UmuC, which in turn activates umuC’s polymerase catalytic activity on damaged DNA.[34] In E. coli, a polymerase «tool belt» model for switching pol III with pol IV at a stalled replication fork, where both polymerases bind simultaneously to the β-clamp, has been proposed.[35] However, the involvement of more than one TLS polymerase working in succession to bypass a lesion has not yet been shown in E. coli. Moreover, Pol IV can catalyze both insertion and extension with high efficiency, whereas pol V is considered the major SOS TLS polymerase. One example is the bypass of intra strand guanine thymine cross-link where it was shown on the basis of the difference in the mutational signatures of the two polymerases, that pol IV and pol V compete for TLS of the intra-strand crosslink.[35]
Family D[edit]

Structures of archaeal polD and eukaryotic Polα. Not only is the general topology conserved, the two also share a bifunctional primase-and-PCNA-binding PIP-box sequence on the C-terminus, similar to both eukaryotic Polα and Polε.[36]
In 1998, the family D of DNA polymerase was discovered in Pyrococcus furiosus and Methanococcus jannaschii.[37] The PolD complex is a heterodimer of two chains, each encoded by DP1 (small proofreading) and DP2 (large catalytic). Unlike other DNA polymerases, the structure and mechanism of the DP2 catalytic core resemble that of multi-subunit RNA polymerases. The DP1-DP2 interface resembles that of Eukaryotic Class B polymerase zinc finger and its small subunit.[17] DP1, a Mre11-like exonuclease,[38] is likely the precursor of small subunit of Pol α and ε, providing proofreading capabilities now lost in Eukaryotes.[25] Its N-terminal HSH domain is similar to AAA proteins, especially Pol III subunit δ and RuvB, in structure.[39] DP2 has a Class II KH domain.[17] Pyrococcus abyssi polD is more heat-stable and more accurate than Taq polymerase, but has not yet been commercialized.[40] It has been proposed that family D DNA polymerase was the first to evolve in cellular organisms and that the replicative polymerase of the Last Universal Cellular Ancestor (LUCA) belonged to family D.[41]
Eukaryotic DNA polymerase[edit]
Polymerases β, λ, σ, μ (beta, lambda, sigma, mu) and TdT[edit]
Family X polymerases contain the well-known eukaryotic polymerase pol β (beta), as well as other eukaryotic polymerases such as Pol σ (sigma), Pol λ (lambda), Pol μ (mu), and Terminal deoxynucleotidyl transferase (TdT). Family X polymerases are found mainly in vertebrates, and a few are found in plants and fungi. These polymerases have highly conserved regions that include two helix-hairpin-helix motifs that are imperative in the DNA-polymerase interactions. One motif is located in the 8 kDa domain that interacts with downstream DNA and one motif is located in the thumb domain that interacts with the primer strand. Pol β, encoded by POLB gene, is required for short-patch base excision repair, a DNA repair pathway that is essential for repairing alkylated or oxidized bases as well as abasic sites. Pol λ and Pol μ, encoded by the POLL and POLM genes respectively, are involved in non-homologous end-joining, a mechanism for rejoining DNA double-strand breaks due to hydrogen peroxide and ionizing radiation, respectively. TdT is expressed only in lymphoid tissue, and adds «n nucleotides» to double-strand breaks formed during V(D)J recombination to promote immunological diversity.[42]
Polymerases α, δ and ε (alpha, delta, and epsilon)[edit]
Pol α (alpha), Pol δ (delta), and Pol ε (epsilon) are members of Family B Polymerases and are the main polymerases involved with nuclear DNA replication. Pol α complex (pol α-DNA primase complex) consists of four subunits: the catalytic subunit POLA1, the regulatory subunit POLA2, and the small and the large primase subunits PRIM1 and PRIM2 respectively. Once primase has created the RNA primer, Pol α starts replication elongating the primer with ~20 nucleotides.[43] Due to its high processivity, Pol δ takes over the leading and lagging strand synthesis from Pol α.[14]: 218–219 Pol δ is expressed by genes POLD1, creating the catalytic subunit, POLD2, POLD3, and POLD4 creating the other subunits that interact with Proliferating Cell Nuclear Antigen (PCNA), which is a DNA clamp that allows Pol δ to possess processivity.[44] Pol ε is encoded by the POLE1, the catalytic subunit, POLE2, and POLE3 gene. It has been reported that the function of Pol ε is to extend the leading strand during replication,[45][46] while Pol δ primarily replicates the lagging strand; however, recent evidence suggested that Pol δ might have a role in replicating the leading strand of DNA as well.[47] Pol ε’s C-terminus «polymerase relic» region, despite being unnecessary for polymerase activity,[48] is thought to be essential to cell vitality. The C-terminus region is thought to provide a checkpoint before entering anaphase, provide stability to the holoenzyme, and add proteins to the holoenzyme necessary for initiation of replication.[49] Pol ε has a larger «palm» domain that provides high processivity independently of PCNA.[48]
Compared to other Family B polymerases, the DEDD exonuclease family responsible for proofreading is inactivated in Pol α.[25] Pol ε is unique in that it has two zinc finger domains and an inactive copy of another family B polymerase in its C-terminal. The presence of this zinc finger has implications in the origins of Eukaryota, which in this case is placed into the Asgard group with archaeal B3 polymerase.[50]
Polymerases η, ι and κ (eta, iota, and kappa)[edit]
Pol η (eta), Pol ι (iota), and Pol κ (kappa), are Family Y DNA polymerases involved in the DNA repair by translation synthesis and encoded by genes POLH, POLI, and POLK respectively. Members of Family Y have five common motifs to aid in binding the substrate and primer terminus and they all include the typical right hand thumb, palm and finger domains with added domains like little finger (LF), polymerase-associated domain (PAD), or wrist. The active site, however, differs between family members due to the different lesions being repaired. Polymerases in Family Y are low-fidelity polymerases, but have been proven to do more good than harm as mutations that affect the polymerase can cause various diseases, such as skin cancer and Xeroderma Pigmentosum Variant (XPS). The importance of these polymerases is evidenced by the fact that gene encoding DNA polymerase η is referred as XPV, because loss of this gene results in the disease Xeroderma Pigmentosum Variant. Pol η is particularly important for allowing accurate translesion synthesis of DNA damage resulting from ultraviolet radiation. The functionality of Pol κ is not completely understood, but researchers have found two probable functions. Pol κ is thought to act as an extender or an inserter of a specific base at certain DNA lesions. All three translesion synthesis polymerases, along with Rev1, are recruited to damaged lesions via stalled replicative DNA polymerases. There are two pathways of damage repair leading researchers to conclude that the chosen pathway depends on which strand contains the damage, the leading or lagging strand.[51]
Polymerases Rev1 and ζ (zeta)[edit]
Pol ζ another B family polymerase, is made of two subunits Rev3, the catalytic subunit, and Rev7 (MAD2L2), which increases the catalytic function of the polymerase, and is involved in translesion synthesis. Pol ζ lacks 3′ to 5′ exonuclease activity, is unique in that it can extend primers with terminal mismatches. Rev1 has three regions of interest in the BRCT domain, ubiquitin-binding domain, and C-terminal domain and has dCMP transferase ability, which adds deoxycytidine opposite lesions that would stall replicative polymerases Pol δ and Pol ε. These stalled polymerases activate ubiquitin complexes that in turn disassociate replication polymerases and recruit Pol ζ and Rev1. Together Pol ζ and Rev1 add deoxycytidine and Pol ζ extends past the lesion. Through a yet undetermined process, Pol ζ disassociates and replication polymerases reassociate and continue replication. Pol ζ and Rev1 are not required for replication, but loss of REV3 gene in budding yeast can cause increased sensitivity to DNA-damaging agents due to collapse of replication forks where replication polymerases have stalled.[52]
Telomerase[edit]
Telomerase is a ribonucleoprotein which functions to replicate ends of linear chromosomes since normal DNA polymerase cannot replicate the ends, or telomeres. The single-strand 3′ overhang of the double-strand chromosome with the sequence 5′-TTAGGG-3′ recruits telomerase. Telomerase acts like other DNA polymerases by extending the 3′ end, but, unlike other DNA polymerases, telomerase does not require a template. The TERT subunit, an example of a reverse transcriptase, uses the RNA subunit to form the primer–template junction that allows telomerase to extend the 3′ end of chromosome ends. The gradual decrease in size of telomeres as the result of many replications over a lifetime are thought to be associated with the effects of aging.[14]: 248–249
Polymerases γ, θ and ν (gamma, theta and nu)[edit]
Pol γ (gamma), Pol θ (theta), and Pol ν (nu) are Family A polymerases. Pol γ, encoded by the POLG gene, was long thought to be the only mitochondrial polymerase. However, recent research shows that at least Pol β (beta), a Family X polymerase, is also present in mitochondria.[53][54] Any mutation that leads to limited or non-functioning Pol γ has a significant effect on mtDNA and is the most common cause of autosomal inherited mitochondrial disorders.[55] Pol γ contains a C-terminus polymerase domain and an N-terminus 3’–5′ exonuclease domain that are connected via the linker region, which binds the accessory subunit. The accessory subunit binds DNA and is required for processivity of Pol γ. Point mutation A467T in the linker region is responsible for more than one-third of all Pol γ-associated mitochondrial disorders.[56] While many homologs of Pol θ, encoded by the POLQ gene, are found in eukaryotes, its function is not clearly understood. The sequence of amino acids in the C-terminus is what classifies Pol θ as Family A polymerase, although the error rate for Pol θ is more closely related to Family Y polymerases. Pol θ extends mismatched primer termini and can bypass abasic sites by adding a nucleotide. It also has Deoxyribophosphodiesterase (dRPase) activity in the polymerase domain and can show ATPase activity in close proximity to ssDNA.[57] Pol ν (nu) is considered to be the least effective of the polymerase enzymes.[58] However, DNA polymerase nu plays an active role in homology repair during cellular responses to crosslinks, fulfilling its role in a complex with helicase.[58]
Plants use two Family A polymerases to copy both the mitochondrial and plastid genomes. They are more similar to bacterial Pol I than they are to mammalian Pol γ.[59]
Reverse transcriptase[edit]
Retroviruses encode an unusual DNA polymerase called reverse transcriptase, which is an RNA-dependent DNA polymerase (RdDp) that synthesizes DNA from a template of RNA. The reverse transcriptase family contain both DNA polymerase functionality and RNase H functionality, which degrades RNA base-paired to DNA. An example of a retrovirus is HIV.[14] Reverse transcriptase is commonly employed in amplification of RNA for research purposes. Using an RNA template, PCR can utilize reverse transcriptase, creating a DNA template. This new DNA template can then be used for typical PCR amplification. The products of such an experiment are thus amplified PCR products from RNA.[9]
Each HIV retrovirus particle contains two RNA genomes, but, after an infection, each virus generates only one provirus.[60] After infection, reverse transcription is accompanied by template switching between the two genome copies (copy choice recombination).[60] From 5 to 14 recombination events per genome occur at each replication cycle.[61] Template switching (recombination) appears to be necessary for maintaining genome integrity and as a repair mechanism for salvaging damaged genomes.[62][60]
Bacteriophage T4 DNA polymerase[edit]
Bacteriophage (phage) T4 encodes a DNA polymerase that catalyzes DNA synthesis in a 5′ to 3′ direction.[63] The phage polymerase also has an exonuclease activity that acts in a 3′ to 5′ direction,[64] and this activity is employed in the proofreading and editing of newly inserted bases.[65] A phage mutant with a temperature sensitive DNA polymerase, when grown at permissive temperatures, was observed to undergo recombination at frequencies that are about two-fold higher than that of wild-type phage.[66]
It was proposed that a mutational alteration in the phage DNA polymerase can stimulate template strand switching (copy choice recombination) during replication.[66]
See also[edit]
- Biological machines
- DNA sequencing
- Enzyme catalysis
- Genetic recombination
- Molecular cloning
- Polymerase chain reaction
- Protein domain dynamics
- Reverse transcription
- RNA polymerase
- Taq DNA polymerase
References[edit]
- ^ Bollum FJ (August 1960). «Calf thymus polymerase». The Journal of Biological Chemistry. 235 (8): 2399–403. doi:10.1016/S0021-9258(18)64634-4. PMID 13802334.
- ^ Falaschi A, Kornberg A (April 1966). «Biochemical studies of bacterial sporulation. II. Deoxy- ribonucleic acid polymerase in spores of Bacillus subtilis». The Journal of Biological Chemistry. 241 (7): 1478–82. doi:10.1016/S0021-9258(18)96736-0. PMID 4957767.
- ^ Lehman IR, Bessman MJ, Simms ES, Kornberg A (July 1958). «Enzymatic synthesis of deoxyribonucleic acid. I. Preparation of substrates and partial purification of an enzyme from Escherichia coli». The Journal of Biological Chemistry. 233 (1): 163–70. doi:10.1016/S0021-9258(19)68048-8. PMID 13563462.
- ^ Richardson CC, Schildkraut CL, Aposhian HV, Kornberg A (January 1964). «Enzymatic synthesis of deoxyribonucleic acid. XIV. Further purification and properties of deoxyribonucleic acid polymerase of Escherichia coli«. The Journal of Biological Chemistry. 239: 222–32. doi:10.1016/S0021-9258(18)51772-5. PMID 14114848.
- ^ Schachman HK, Adler J, Radding CM, Lehman IR, Kornberg A (November 1960). «Enzymatic synthesis of deoxyribonucleic acid. VII. Synthesis of a polymer of deoxyadenylate and deoxythymidylate». The Journal of Biological Chemistry. 235 (11): 3242–9. doi:10.1016/S0021-9258(20)81345-3. PMID 13747134.
- ^ Zimmerman BK (May 1966). «Purification and properties of deoxyribonucleic acid polymerase from Micrococcus lysodeikticus». The Journal of Biological Chemistry. 241 (9): 2035–41. doi:10.1016/S0021-9258(18)96662-7. PMID 5946628.
- ^ «The Nobel Prize in Physiology or Medicine 1959». Nobel Foundation. Retrieved December 1, 2012.
- ^ Tessman I, Kennedy MA (February 1994). «DNA polymerase II of Escherichia coli in the bypass of abasic sites in vivo». Genetics. 136 (2): 439–48. doi:10.1093/genetics/136.2.439. PMC 1205799. PMID 7908652.
- ^ a b Lehninger AL, Cox MM, Nelson DL (2013). Lehninger principles of biochemistry (6th ed.). New York: W.H. Freeman and Company. ISBN 978-1-4292-3414-6. OCLC 824794893.
- ^ Garrett G (2013). Biochemistry. Mary Finch.
- ^ Hunter WN, Brown T, Anand NN, Kennard O (1986). «Structure of an adenine-cytosine base pair in DNA and its implications for mismatch repair». Nature. 320 (6062): 552–5. Bibcode:1986Natur.320..552H. doi:10.1038/320552a0. PMID 3960137. S2CID 4319887.
- ^ Swan MK, Johnson RE, Prakash L, Prakash S, Aggarwal AK (September 2009). «Structural basis of high-fidelity DNA synthesis by yeast DNA polymerase delta». Nature Structural & Molecular Biology. 16 (9): 979–86. doi:10.1038/nsmb.1663. PMC 3055789. PMID 19718023.
- ^ Steitz TA (June 1999). «DNA polymerases: structural diversity and common mechanisms». The Journal of Biological Chemistry. 274 (25): 17395–8. doi:10.1074/jbc.274.25.17395. PMID 10364165.
- ^ a b c d e Losick R, Watson JD, Baker TA, Bell S, Gann A, Levine MW (2008). Molecular biology of the gene (6th ed.). San Francisco: Pearson/Benjamin Cummings. ISBN 978-0-8053-9592-1.
- ^ McCarthy D, Minner C, Bernstein H, Bernstein C (October 1976). «DNA elongation rates and growing point distributions of wild-type phage T4 and a DNA-delay amber mutant». Journal of Molecular Biology. 106 (4): 963–81. doi:10.1016/0022-2836(76)90346-6. PMID 789903.
- ^ Filée J, Forterre P, Sen-Lin T, Laurent J (June 2002). «Evolution of DNA polymerase families: evidences for multiple gene exchange between cellular and viral proteins» (PDF). Journal of Molecular Evolution. 54 (6): 763–73. Bibcode:2002JMolE..54..763F. CiteSeerX 10.1.1.327.4738. doi:10.1007/s00239-001-0078-x. PMID 12029358. S2CID 15852365. Archived from the original (PDF) on 2020-07-29. Retrieved 2019-09-23.
- ^ a b c Raia P, Carroni M, Henry E, Pehau-Arnaudet G, Brûlé S, Béguin P, Henneke G, Lindahl E, Delarue M, Sauguet L (January 2019). «Structure of the DP1-DP2 PolD complex bound with DNA and its implications for the evolutionary history of DNA and RNA polymerases». PLOS Biology. 17 (1): e3000122. doi:10.1371/journal.pbio.3000122. PMC 6355029. PMID 30657780.
- ^ Boehm EM, Powers KT, Kondratick CM, Spies M, Houtman JC, Washington MT (April 2016). «The Proliferating Cell Nuclear Antigen (PCNA)-interacting Protein (PIP) Motif of DNA Polymerase η Mediates Its Interaction with the C-terminal Domain of Rev1». The Journal of Biological Chemistry. 291 (16): 8735–44. doi:10.1074/jbc.M115.697938. PMC 4861442. PMID 26903512.
- ^ Yang W (May 2014). «An overview of Y-Family DNA polymerases and a case study of human DNA polymerase η». Biochemistry. 53 (17): 2793–803. doi:10.1021/bi500019s. PMC 4018060. PMID 24716551.
- ^ Maga G, Hubscher U, Spadari S, Villani G (2010). DNA Polymerases: Discovery, Characterization Functions in Cellular DNA Transactions. World Scientific Publishing Company. ISBN 978-981-4299-16-9.
- ^ Choi CH, Burton ZF, Usheva A (February 2004). «Auto-acetylation of transcription factors as a control mechanism in gene expression». Cell Cycle. 3 (2): 114–5. doi:10.4161/cc.3.2.651. PMID 14712067.
- ^ Chien A, Edgar DB, Trela JM (September 1976). «Deoxyribonucleic acid polymerase from the extreme thermophile Thermus aquaticus». Journal of Bacteriology. 127 (3): 1550–7. doi:10.1128/JB.127.3.1550-1557.1976. PMC 232952. PMID 8432.
- ^ a b Banach-Orlowska M, Fijalkowska IJ, Schaaper RM, Jonczyk P (October 2005). «DNA polymerase II as a fidelity factor in chromosomal DNA synthesis in Escherichia coli». Molecular Microbiology. 58 (1): 61–70. doi:10.1111/j.1365-2958.2005.04805.x. PMID 16164549. S2CID 12002486.
- ^ InterPro protein view: P61875
- ^ a b c Makarova KS, Krupovic M, Koonin EV (2014). «Evolution of replicative DNA polymerases in archaea and their contributions to the eukaryotic replication machinery». Frontiers in Microbiology. 5: 354. doi:10.3389/fmicb.2014.00354. PMC 4104785. PMID 25101062.
- ^ Rohe M, Schrage K, Meinhardt F (December 1991). «The linear plasmid pMC3-2 from Morchella conica is structurally related to adenoviruses». Current Genetics. 20 (6): 527–33. doi:10.1007/BF00334782. PMID 1782679. S2CID 35072924.
- ^ Olson MW, Dallmann HG, McHenry CS (December 1995). «DnaX complex of Escherichia coli DNA polymerase III holoenzyme. The chi psi complex functions by increasing the affinity of tau and gamma for delta.delta’ to a physiologically relevant range». The Journal of Biological Chemistry. 270 (49): 29570–7. doi:10.1074/jbc.270.49.29570. PMID 7494000.
- ^ Liao Y, Li Y, Schroeder JW, Simmons LA, Biteen JS (December 2016). «Single-Molecule DNA Polymerase Dynamics at a Bacterial Replisome in Live Cells». Biophysical Journal. 111 (12): 2562–2569. Bibcode:2016BpJ…111.2562L. doi:10.1016/j.bpj.2016.11.006. PMC 5192695. PMID 28002733.
- ^ a b c Xu ZQ, Dixon NE (December 2018). «Bacterial replisomes». Current Opinion in Structural Biology. 53: 159–168. doi:10.1016/j.sbi.2018.09.006. PMID 30292863.
- ^ Goodman MF (2002). «Error-prone repair DNA polymerases in prokaryotes and eukaryotes». Annual Review of Biochemistry. 71: 17–50. doi:10.1146/annurev.biochem.71.083101.124707. PMID 12045089. S2CID 1979429.
- ^ Mori T, Nakamura T, Okazaki N, Furukohri A, Maki H, Akiyama MT (2012). «Escherichia coli DinB inhibits replication fork progression without significantly inducing the SOS response». Genes & Genetic Systems. 87 (2): 75–87. doi:10.1266/ggs.87.75. PMID 22820381.
- ^ Jarosz DF, Godoy VG, Walker GC (April 2007). «Proficient and accurate bypass of persistent DNA lesions by DinB DNA polymerases». Cell Cycle. 6 (7): 817–22. doi:10.4161/cc.6.7.4065. PMID 17377496.
- ^ Patel M, Jiang Q, Woodgate R, Cox MM, Goodman MF (June 2010). «A new model for SOS-induced mutagenesis: how RecA protein activates DNA polymerase V». Critical Reviews in Biochemistry and Molecular Biology. 45 (3): 171–84. doi:10.3109/10409238.2010.480968. PMC 2874081. PMID 20441441.
- ^ Sutton MD, Walker GC (July 2001). «Managing DNA polymerases: coordinating DNA replication, DNA repair, and DNA recombination». Proceedings of the National Academy of Sciences of the United States of America. 98 (15): 8342–9. Bibcode:2001PNAS…98.8342S. doi:10.1073/pnas.111036998. PMC 37441. PMID 11459973.
- ^ a b Raychaudhury P, Basu AK (March 2011). «Genetic requirement for mutagenesis of the G[8,5-Me]T cross-link in Escherichia coli: DNA polymerases IV and V compete for error-prone bypass». Biochemistry. 50 (12): 2330–8. doi:10.1021/bi102064z. PMC 3062377. PMID 21302943.
- ^ Madru C, Henneke G, Raia P, Hugonneau-Beaufet I, Pehau-Arnaudet G, England P, et al. (March 2020). «Structural basis for the increased processivity of D-family DNA polymerases in complex with PCNA». Nature Communications. 11 (1): 1591. doi:10.1038/s41467-020-15392-9. PMID 32221299.
- ^ Ishino Y, Komori K, Cann IK, Koga Y (April 1998). «A novel DNA polymerase family found in Archaea». Journal of Bacteriology. 180 (8): 2232–6. doi:10.1128/JB.180.8.2232-2236.1998. PMC 107154. PMID 9555910.
- ^ Sauguet L, Raia P, Henneke G, Delarue M (2016). «Shared active site architecture between archaeal PolD and multi-subunit RNA polymerases revealed by X-ray crystallography». Nature Communications. 7: 12227. Bibcode:2016NatCo…712227S. doi:10.1038/ncomms12227. PMC 4996933. PMID 27548043.
- ^ Yamasaki K, Urushibata Y, Yamasaki T, Arisaka F, Matsui I (August 2010). «Solution structure of the N-terminal domain of the archaeal D-family DNA polymerase small subunit reveals evolutionary relationship to eukaryotic B-family polymerases». FEBS Letters. 584 (15): 3370–5. doi:10.1016/j.febslet.2010.06.026. PMID 20598295. S2CID 11229530.
- ^ Ishino S, Ishino Y (2014). «DNA polymerases as useful reagents for biotechnology — the history of developmental research in the field». Frontiers in Microbiology. 5: 465. doi:10.3389/fmicb.2014.00465. PMC 4148896. PMID 25221550.
- ^ Koonin EV, Krupovic M, Ishino S, Ishino Y (June 2020). «The replication machinery of LUCA: common origin of DNA replication and transcription». BMC Biology. 18 (1): 61. doi:10.1186/s12915-020-00800-9. PMC 7281927. PMID 32517760.
- ^ Yamtich J, Sweasy JB (May 2010). «DNA polymerase family X: function, structure, and cellular roles». Biochimica et Biophysica Acta (BBA) — Proteins and Proteomics. 1804 (5): 1136–50. doi:10.1016/j.bbapap.2009.07.008. PMC 2846199. PMID 19631767.
- ^ Chansky ML, Marks A, Peet A (2012). Marks’ Basic Medical Biochemistry: a clinical approach (4th ed.). Philadelphia: Wolter Kluwer Health/Lippincott Williams & Wilkins. p. chapter13. ISBN 978-1608315727.
- ^ Chung DW, Zhang JA, Tan CK, Davie EW, So AG, Downey KM (December 1991). «Primary structure of the catalytic subunit of human DNA polymerase delta and chromosomal location of the gene». Proceedings of the National Academy of Sciences of the United States of America. 88 (24): 11197–201. Bibcode:1991PNAS…8811197C. doi:10.1073/pnas.88.24.11197. PMC 53101. PMID 1722322.
- ^ Pursell ZF, Isoz I, Lundström EB, Johansson E, Kunkel TA (July 2007). «Yeast DNA polymerase epsilon participates in leading-strand DNA replication». Science. 317 (5834): 127–30. Bibcode:2007Sci…317..127P. doi:10.1126/science.1144067. PMC 2233713. PMID 17615360.
- ^ Lujan SA, Williams JS, Kunkel TA (September 2016). «DNA Polymerases Divide the Labor of Genome Replication». Trends in Cell Biology. 26 (9): 640–654. doi:10.1016/j.tcb.2016.04.012. PMC 4993630. PMID 27262731.
- ^ Johnson RE, Klassen R, Prakash L, Prakash S (July 2015). «A Major Role of DNA Polymerase δ in Replication of Both the Leading and Lagging DNA Strands». Molecular Cell. 59 (2): 163–175. doi:10.1016/j.molcel.2015.05.038. PMC 4517859. PMID 26145172.
- ^ a b Doublié S, Zahn KE (2014). «Structural insights into eukaryotic DNA replication». Frontiers in Microbiology. 5: 444. doi:10.3389/fmicb.2014.00444. PMC 4142720. PMID 25202305.
- ^ Edwards S, Li CM, Levy DL, Brown J, Snow PM, Campbell JL (April 2003). «Saccharomyces cerevisiae DNA polymerase epsilon and polymerase sigma interact physically and functionally, suggesting a role for polymerase epsilon in sister chromatid cohesion». Molecular and Cellular Biology. 23 (8): 2733–48. doi:10.1128/mcb.23.8.2733-2748.2003. PMC 152548. PMID 12665575.
- ^ Zaremba-Niedzwiedzka K, Caceres EF, Saw JH, Bäckström D, Juzokaite L, Vancaester E, Seitz KW, Anantharaman K, Starnawski P, Kjeldsen KU, Stott MB, Nunoura T, Banfield JF, Schramm A, Baker BJ, Spang A, Ettema TJ (January 2017). «Asgard archaea illuminate the origin of eukaryotic cellular complexity». Nature. 541 (7637): 353–358. Bibcode:2017Natur.541..353Z. doi:10.1038/nature21031. OSTI 1580084. PMID 28077874. S2CID 4458094.
- ^ Ohmori H, Hanafusa T, Ohashi E, Vaziri C (2009). Separate roles of structured and unstructured regions of Y-family DNA polymerases. Advances in Protein Chemistry and Structural Biology. Vol. 78. pp. 99–146. doi:10.1016/S1876-1623(08)78004-0. ISBN 9780123748270. PMC 3103052. PMID 20663485.
- ^ Gan GN, Wittschieben JP, Wittschieben BØ, Wood RD (January 2008). «DNA polymerase zeta (pol zeta) in higher eukaryotes». Cell Research. 18 (1): 174–83. doi:10.1038/cr.2007.117. PMID 18157155.
- ^ Bienstock R, Beard W, Wilson S (August 2014). «Phylogenetic analysis and evolutionary origins of DNA polymerase X-family members». DNA Repair. 22: 77–88. doi:10.1016/j.dnarep.2014.07.003. PMC 4260717. PMID 25112931.
- ^ Prasad R, et al. (October 2017). «DNA polymerase β: A missing link of the base excision repair machinery in mammalian mitochondria». DNA Repair. 60: 77–88. doi:10.1016/j.dnarep.2017.10.011. PMC 5919216. PMID 29100041.
- ^ Zhang L, Chan SS, Wolff DJ (July 2011). «Mitochondrial disorders of DNA polymerase γ dysfunction: from anatomic to molecular pathology diagnosis». Archives of Pathology & Laboratory Medicine. 135 (7): 925–34. doi:10.5858/2010-0356-RAR.1. PMC 3158670. PMID 21732785.
- ^ Stumpf JD, Copeland WC (January 2011). «Mitochondrial DNA replication and disease: insights from DNA polymerase γ mutations». Cellular and Molecular Life Sciences. 68 (2): 219–33. doi:10.1007/s00018-010-0530-4. PMC 3046768. PMID 20927567.
- ^ Hogg M, Sauer-Eriksson AE, Johansson E (March 2012). «Promiscuous DNA synthesis by human DNA polymerase θ». Nucleic Acids Research. 40 (6): 2611–22. doi:10.1093/nar/gkr1102. PMC 3315306. PMID 22135286.
- ^ a b «UniProtKB — Q7Z5Q5 (DPOLN_HUMAN)». Uniprot. Retrieved 5 July 2018.
- ^ Cupp JD, Nielsen BL (November 2014). «Minireview: DNA replication in plant mitochondria». Mitochondrion. 19 Pt B: 231–7. doi:10.1016/j.mito.2014.03.008. PMC 417701. PMID 24681310.
- ^ a b c Rawson JM, Nikolaitchik OA, Keele BF, Pathak VK, Hu WS (November 2018). «Recombination is required for efficient HIV-1 replication and the maintenance of viral genome integrity». Nucleic Acids Research. 46 (20): 10535–10545. doi:10.1093/nar/gky910. PMC 6237782. PMID 30307534.
- ^ Cromer D, Grimm AJ, Schlub TE, Mak J, Davenport MP (January 2016). «Estimating the in-vivo HIV template switching and recombination rate». AIDS. 30 (2): 185–92. doi:10.1097/QAD.0000000000000936. PMID 26691546. S2CID 20086739.
- ^ Hu WS, Temin HM (November 1990). «Retroviral recombination and reverse transcription». Science. 250 (4985): 1227–33. Bibcode:1990Sci…250.1227H. doi:10.1126/science.1700865. PMID 1700865.
- ^ Goulian M, Lucas ZJ, Kornberg A (February 1968). «Enzymatic synthesis of deoxyribonucleic acid. XXV. Purification and properties of deoxyribonucleic acid polymerase induced by infection with phage T4». The Journal of Biological Chemistry. 243 (3): 627–38. doi:10.1016/S0021-9258(18)93650-1. PMID 4866523.
- ^ Huang WM, Lehman IR (May 1972). «On the exonuclease activity of phage T4 deoxyribonucleic acid polymerase». The Journal of Biological Chemistry. 247 (10): 3139–46. doi:10.1016/S0021-9258(19)45224-1. PMID 4554914.
- ^ Gillin FD, Nossal NG (September 1976). «Control of mutation frequency by bacteriophage T4 DNA polymerase. I. The CB120 antimutator DNA polymerase is defective in strand displacement». The Journal of Biological Chemistry. 251 (17): 5219–24. doi:10.1016/S0021-9258(17)33149-6. PMID 956182.
- ^ a b Bernstein H (August 1967). «The effect on recombination of mutational defects in the DNA-polymerase and deoxycytidylate hydroxymethylase of phage T4D». Genetics. 56 (4): 755–69. doi:10.1093/genetics/56.4.755. PMC 1211652. PMID 6061665.
Further reading[edit]
- Burgers PM, Koonin EV, Bruford E, Blanco L, Burtis KC, Christman MF, Copeland WC, Friedberg EC, Hanaoka F, Hinkle DC, Lawrence CW, Nakanishi M, Ohmori H, Prakash L, Prakash S, Reynaud CA, Sugino A, Todo T, Wang Z, Weill JC, Woodgate R (November 2001). «Eukaryotic DNA polymerases: proposal for a revised nomenclature». The Journal of Biological Chemistry. 276 (47): 43487–90. doi:10.1074/jbc.R100056200. PMID 11579108.
External links[edit]
- DNA+polymerases at the US National Library of Medicine Medical Subject Headings (MeSH)
- PDB Molecule of the Month DNA polymerase
- Unusual repair mechanism in DNA polymerase lambda, Ohio State University, July 25, 2006.
- A great animation of DNA Polymerase from WEHI at 1:45 minutes in
- 3D macromolecular structures of DNA polymerase from the EM Data Bank(EMDB)
| DNA-directed DNA polymerase | |||||||
|---|---|---|---|---|---|---|---|

3D structure of the DNA-binding helix-turn-helix motifs in human DNA polymerase beta (based on PDB file 7ICG) |
|||||||
| Identifiers | |||||||
| EC no. | 2.7.7.7 | ||||||
| CAS no. | 9012-90-2 | ||||||
| Databases | |||||||
| IntEnz | IntEnz view | ||||||
| BRENDA | BRENDA entry | ||||||
| ExPASy | NiceZyme view | ||||||
| KEGG | KEGG entry | ||||||
| MetaCyc | metabolic pathway | ||||||
| PRIAM | profile | ||||||
| PDB structures | RCSB PDB PDBe PDBsum | ||||||
| Gene Ontology | AmiGO / QuickGO | ||||||
|
A DNA polymerase is a member of a family of enzymes that catalyze the synthesis of DNA molecules from nucleoside triphosphates, the molecular precursors of DNA. These enzymes are essential for DNA replication and usually work in groups to create two identical DNA duplexes from a single original DNA duplex. During this process, DNA polymerase «reads» the existing DNA strands to create two new strands that match the existing ones.[1][2][3][4][5][6]
These enzymes catalyze the chemical reaction
- deoxynucleoside triphosphate + DNAn ⇌ pyrophosphate + DNAn+1.
DNA polymerase adds nucleotides to the three prime (3′)-end of a DNA strand, one nucleotide at a time. Every time a cell divides, DNA polymerases are required to duplicate the cell’s DNA, so that a copy of the original DNA molecule can be passed to each daughter cell. In this way, genetic information is passed down from generation to generation.
Before replication can take place, an enzyme called helicase unwinds the DNA molecule from its tightly woven form, in the process breaking the hydrogen bonds between the nucleotide bases. This opens up or «unzips» the double-stranded DNA to give two single strands of DNA that can be used as templates for replication in the above reaction.
History[edit]
In 1956, Arthur Kornberg and colleagues discovered DNA polymerase I (Pol I), in Escherichia coli. They described the DNA replication process by which DNA polymerase copies the base sequence of a template DNA strand. Kornberg was later awarded the Nobel Prize in Physiology or Medicine in 1959 for this work.[7] DNA polymerase II was discovered by Thomas Kornberg (the son of Arthur Kornberg) and Malcolm E. Gefter in 1970 while further elucidating the role of Pol I in E. coli DNA replication.[8] Three more DNA polymerases have been found in E. coli, including DNA polymerase III (discovered in the 1970s) and DNA polymerases IV and V (discovered in 1999).[9]
Function[edit]

DNA polymerase moves along the old strand in the 3’–5′ direction, creating a new strand having a 5’–3′ direction.

DNA polymerase with proofreading ability
The main function of DNA polymerase is to synthesize DNA from deoxyribonucleotides, the building blocks of DNA. The DNA copies are created by the pairing of nucleotides to bases present on each strand of the original DNA molecule. This pairing always occurs in specific combinations, with cytosine along with guanine, and thymine along with adenine, forming two separate pairs, respectively. By contrast, RNA polymerases synthesize RNA from ribonucleotides from either RNA or DNA.
When synthesizing new DNA, DNA polymerase can add free nucleotides only to the 3′ end of the newly forming strand. This results in elongation of the newly forming strand in a 5’–3′ direction.
It is important to note that the directionality of the newly forming strand (the daughter strand) is opposite to the direction in which DNA polymerase moves along the template strand. Since DNA polymerase requires a free 3′ OH group for initiation of synthesis, it can synthesize in only one direction by extending the 3′ end of the preexisting nucleotide chain. Hence, DNA polymerase moves along the template strand in a 3’–5′ direction, and the daughter strand is formed in a 5’–3′ direction. This difference enables the resultant double-strand DNA formed to be composed of two DNA strands that are antiparallel to each other.
The function of DNA polymerase is not quite perfect, with the enzyme making about one mistake for every billion base pairs copied. Error correction is a property of some, but not all DNA polymerases. This process corrects mistakes in newly synthesized DNA. When an incorrect base pair is recognized, DNA polymerase moves backwards by one base pair of DNA. The 3’–5′ exonuclease activity of the enzyme allows the incorrect base pair to be excised (this activity is known as proofreading). Following base excision, the polymerase can re-insert the correct base and replication can continue forwards. This preserves the integrity of the original DNA strand that is passed onto the daughter cells.
Fidelity is very important in DNA replication. Mismatches in DNA base pairing can potentially result in dysfunctional proteins and could lead to cancer. Many DNA polymerases contain an exonuclease domain, which acts in detecting base pair mismatches and further performs in the removal of the incorrect nucleotide to be replaced by the correct one.[10] The shape and the interactions accommodating the Watson and Crick base pair are what primarily contribute to the detection or error. Hydrogen bonds play a key role in base pair binding and interaction. The loss of an interaction, which occurs at a mismatch, is said to trigger a shift in the balance, for the binding of the template-primer, from the polymerase, to the exonuclease domain. In addition, an incorporation of a wrong nucleotide causes a retard in DNA polymerization. This delay gives time for the DNA to be switched from the polymerase site to the exonuclease site. Different conformational changes and loss of interaction occur at different mismatches. In a purine:pyrimidine mismatch there is a displacement of the pyrimidine towards the major groove and the purine towards the minor groove. Relative to the shape of DNA polymerase’s binding pocket, steric clashes occur between the purine and residues in the minor groove, and important van der Waals and electrostatic interactions are lost by the pyrimidine.[11] Pyrimidine:pyrimidine and purine:purine mismatches present less notable changes since the bases are displaced towards the major groove, and less steric hindrance is experienced. However, although the different mismatches result in different steric properties, DNA polymerase is still able to detect and differentiate them so uniformly and maintain fidelity in DNA replication.[12] DNA polymerization is also critical for many mutagenesis processes and is widely employed in biotechnologies.
Structure[edit]
The known DNA polymerases have highly conserved structure, which means that their overall catalytic subunits vary very little from species to species, independent of their domain structures. Conserved structures usually indicate important, irreplaceable functions of the cell, the maintenance of which provides evolutionary advantages. The shape can be described as resembling a right hand with thumb, finger, and palm domains. The palm domain appears to function in catalyzing the transfer of phosphoryl groups in the phosphoryl transfer reaction. DNA is bound to the palm when the enzyme is active. This reaction is believed to be catalyzed by a two-metal-ion mechanism. The finger domain functions to bind the nucleoside triphosphates with the template base. The thumb domain plays a potential role in the processivity, translocation, and positioning of the DNA.[13]
Processivity[edit]
DNA polymerase’s rapid catalysis is due to its processive nature. Processivity is a characteristic of enzymes that function on polymeric substrates. In the case of DNA polymerase, the degree of processivity refers to the average number of nucleotides added each time the enzyme binds a template. The average DNA polymerase requires about one second locating and binding a primer/template junction. Once it is bound, a nonprocessive DNA polymerase adds nucleotides at a rate of one nucleotide per second.[14]: 207–208 Processive DNA polymerases, however, add multiple nucleotides per second, drastically increasing the rate of DNA synthesis. The degree of processivity is directly proportional to the rate of DNA synthesis. The rate of DNA synthesis in a living cell was first determined as the rate of phage T4 DNA elongation in phage infected E. coli. During the period of exponential DNA increase at 37 °C, the rate was 749 nucleotides per second.[15]
DNA polymerase’s ability to slide along the DNA template allows increased processivity. There is a dramatic increase in processivity at the replication fork. This increase is facilitated by the DNA polymerase’s association with proteins known as the sliding DNA clamp. The clamps are multiple protein subunits associated in the shape of a ring. Using the hydrolysis of ATP, a class of proteins known as the sliding clamp loading proteins open up the ring structure of the sliding DNA clamps allowing binding to and release from the DNA strand. Protein–protein interaction with the clamp prevents DNA polymerase from diffusing from the DNA template, thereby ensuring that the enzyme binds the same primer/template junction and continues replication.[14]: 207–208 DNA polymerase changes conformation, increasing affinity to the clamp when associated with it and decreasing affinity when it completes the replication of a stretch of DNA to allow release from the clamp.
Variation across species[edit]
| DNA polymerase family A | |||||||
|---|---|---|---|---|---|---|---|

c:o6-methyl-guanine pair in the polymerase-2 basepair position |
|||||||
| Identifiers | |||||||
| Symbol | DNA_pol_A | ||||||
| Pfam | PF00476 | ||||||
| InterPro | IPR001098 | ||||||
| SMART | — | ||||||
| PROSITE | PDOC00412 | ||||||
| SCOP2 | 1dpi / SCOPe / SUPFAM | ||||||
|
| DNA polymerase family B | |||||||
|---|---|---|---|---|---|---|---|

crystal structure of rb69 gp43 in complex with dna containing thymine glycol |
|||||||
| Identifiers | |||||||
| Symbol | DNA_pol_B | ||||||
| Pfam | PF00136 | ||||||
| Pfam clan | CL0194 | ||||||
| InterPro | IPR006134 | ||||||
| PROSITE | PDOC00107 | ||||||
| SCOP2 | 1noy / SCOPe / SUPFAM | ||||||
|
| DNA polymerase type B, organellar and viral | |||||||
|---|---|---|---|---|---|---|---|

phi29 dna polymerase, orthorhombic crystal form, ssdna complex |
|||||||
| Identifiers | |||||||
| Symbol | DNA_pol_B_2 | ||||||
| Pfam | PF03175 | ||||||
| Pfam clan | CL0194 | ||||||
| InterPro | IPR004868 | ||||||
|
Based on sequence homology, DNA polymerases can be further subdivided into seven different families: A, B, C, D, X, Y, and RT.
Some viruses also encode special DNA polymerases, such as Hepatitis B virus DNA polymerase. These may selectively replicate viral DNA through a variety of mechanisms. Retroviruses encode an unusual DNA polymerase called reverse transcriptase, which is an RNA-dependent DNA polymerase (RdDp). It polymerizes DNA from a template of RNA.
| Family[16] | Types of DNA polymerase | Taxa | Examples | Feature |
|---|---|---|---|---|
| A | Replicative and Repair Polymerases | Eukaryotic and Prokaryotic | T7 DNA polymerase, Pol I, Pol γ, θ, and ν | Two exonuclease domains (3′-5′ and 5′-3′) |
| B | Replicative and Repair Polymerases | Eukaryotic and Prokaryotic | Pol II, Pol B, Pol ζ, Pol α, δ, and ε | 3′-5 exonuclease (proofreading); viral ones use protein primer |
| C | Replicative Polymerases | Prokaryotic | Pol III | 3′-5 exonuclease (proofreading) |
| D | Replicative Polymerases | Euryarchaeota | PolD (DP1/DP2 heterodimer)[17] | No «hand» feature, double barrel RNA polymerase-like; 3′-5 exonuclease (proofreading) |
| X | Replicative and Repair Polymerases | Eukaryotic | Pol β, Pol σ, Pol λ, Pol μ, and terminal deoxynucleotidyl transferase | template optional; 5′ phosphatase (only Pol β); weak «hand» feature |
| Y | Replicative and Repair Polymerases | Eukaryotic and Prokaryotic | Pol ι, Pol κ, Pol η,[18] Pol IV, and Pol V | Translesion synthesis[19] |
| RT | Replicative and Repair Polymerases | Viruses, Retroviruses, and Eukaryotic | Telomerase, Hepatitis B virus | RNA-dependent |
Prokaryotic polymerase[edit]
Prokaryotic polymerases exist in two forms: core polymerase and holoenzyme. Core polymerase synthesizes DNA from the DNA template but it cannot initiate the synthesis alone or accurately. Holoenzyme accurately initiates synthesis.
Pol I[edit]
Prokaryotic family A polymerases include the DNA polymerase I (Pol I) enzyme, which is encoded by the polA gene and ubiquitous among prokaryotes. This repair polymerase is involved in excision repair with both 3’–5′ and 5’–3′ exonuclease activity and processing of Okazaki fragments generated during lagging strand synthesis.[20] Pol I is the most abundant polymerase, accounting for >95% of polymerase activity in E. coli; yet cells lacking Pol I have been found suggesting Pol I activity can be replaced by the other four polymerases. Pol I adds ~15-20 nucleotides per second, thus showing poor processivity. Instead, Pol I starts adding nucleotides at the RNA primer:template junction known as the origin of replication (ori). Approximately 400 bp downstream from the origin, the Pol III holoenzyme is assembled and takes over replication at a highly processive speed and nature.[21]
Taq polymerase is a heat-stable enzyme of this family that lacks proofreading ability.[22]
Pol II[edit]
DNA polymerase II is a family B polymerase encoded by the polB gene. Pol II has 3’–5′ exonuclease activity and participates in DNA repair, replication restart to bypass lesions, and its cell presence can jump from ~30-50 copies per cell to ~200–300 during SOS induction. Pol II is also thought to be a backup to Pol III as it can interact with holoenzyme proteins and assume a high level of processivity. The main role of Pol II is thought to be the ability to direct polymerase activity at the replication fork and help stalled Pol III bypass terminal mismatches.[23]
Pfu DNA polymerase is a heat-stable enzyme of this family found in the hyperthermophilic archaeon Pyrococcus furiosus.[24] Detailed classification divides family B in archaea into B1, B2, B3, in which B2 is a group of pseudoenzymes. Pfu belongs to family B3. Others PolBs found in archaea are part of «Casposons», Cas1-dependent transposons.[25] Some viruses (including Φ29 DNA polymerase) and mitochondrial plasmids carry polB as well.[26]
Pol III[edit]
DNA polymerase III holoenzyme is the primary enzyme involved in DNA replication in E. coli and belongs to family C polymerases. It consists of three assemblies: the pol III core, the beta sliding clamp processivity factor, and the clamp-loading complex. The core consists of three subunits: α, the polymerase activity hub, ɛ, exonucleolytic proofreader, and θ, which may act as a stabilizer for ɛ. The beta sliding clamp processivity factor is also present in duplicate, one for each core, to create a clamp that encloses DNA allowing for high processivity.[27] The third assembly is a seven-subunit (τ2γδδ′χψ) clamp loader complex.
The old textbook «trombone model» depicts an elongation complex with two equivalents of the core enzyme at each replication fork (RF), one for each strand, the lagging and leading.[23] However, recent evidence from single-molecule studies indicates an average of three stoichiometric equivalents of core enzyme at each RF for both Pol III and its counterpart in B. subtilis, PolC.[28] In-cell fluorescent microscopy has revealed that leading strand synthesis may not be completely continuous, and Pol III* (i.e., the holoenzyme α, ε, τ, δ and χ subunits without the ß2 sliding clamp) has a high frequency of dissociation from active RFs.[29] In these studies, the replication fork turnover rate was about 10s for Pol III*, 47s for the ß2 sliding clamp, and 15m for the DnaB helicase. This suggests that the DnaB helicase may remain stably associated at RFs and serve as a nucleation point for the competent holoenzyme. In vitro single-molecule studies have shown that Pol III* has a high rate of RF turnover when in excess, but remains stably associated with replication forks when concentration is limiting.[29] Another single-molecule study showed that DnaB helicase activity and strand elongation can proceed with decoupled, stochastic kinetics.[29]
Pol IV[edit]
In E. coli, DNA polymerase IV (Pol IV) is an error-prone DNA polymerase involved in non-targeted mutagenesis.[30] Pol IV is a Family Y polymerase expressed by the dinB gene that is switched on via SOS induction caused by stalled polymerases at the replication fork. During SOS induction, Pol IV production is increased tenfold and one of the functions during this time is to interfere with Pol III holoenzyme processivity. This creates a checkpoint, stops replication, and allows time to repair DNA lesions via the appropriate repair pathway.[31] Another function of Pol IV is to perform translesion synthesis at the stalled replication fork like, for example, bypassing N2-deoxyguanine adducts at a faster rate than transversing undamaged DNA. Cells lacking the dinB gene have a higher rate of mutagenesis caused by DNA damaging agents.[32]
Pol V[edit]
DNA polymerase V (Pol V) is a Y-family DNA polymerase that is involved in SOS response and translesion synthesis DNA repair mechanisms.[33] Transcription of Pol V via the umuDC genes is highly regulated to produce only Pol V when damaged DNA is present in the cell generating an SOS response. Stalled polymerases causes RecA to bind to the ssDNA, which causes the LexA protein to autodigest. LexA then loses its ability to repress the transcription of the umuDC operon. The same RecA-ssDNA nucleoprotein posttranslationally modifies the UmuD protein into UmuD’ protein. UmuD and UmuD’ form a heterodimer that interacts with UmuC, which in turn activates umuC’s polymerase catalytic activity on damaged DNA.[34] In E. coli, a polymerase «tool belt» model for switching pol III with pol IV at a stalled replication fork, where both polymerases bind simultaneously to the β-clamp, has been proposed.[35] However, the involvement of more than one TLS polymerase working in succession to bypass a lesion has not yet been shown in E. coli. Moreover, Pol IV can catalyze both insertion and extension with high efficiency, whereas pol V is considered the major SOS TLS polymerase. One example is the bypass of intra strand guanine thymine cross-link where it was shown on the basis of the difference in the mutational signatures of the two polymerases, that pol IV and pol V compete for TLS of the intra-strand crosslink.[35]
Family D[edit]

Structures of archaeal polD and eukaryotic Polα. Not only is the general topology conserved, the two also share a bifunctional primase-and-PCNA-binding PIP-box sequence on the C-terminus, similar to both eukaryotic Polα and Polε.[36]
In 1998, the family D of DNA polymerase was discovered in Pyrococcus furiosus and Methanococcus jannaschii.[37] The PolD complex is a heterodimer of two chains, each encoded by DP1 (small proofreading) and DP2 (large catalytic). Unlike other DNA polymerases, the structure and mechanism of the DP2 catalytic core resemble that of multi-subunit RNA polymerases. The DP1-DP2 interface resembles that of Eukaryotic Class B polymerase zinc finger and its small subunit.[17] DP1, a Mre11-like exonuclease,[38] is likely the precursor of small subunit of Pol α and ε, providing proofreading capabilities now lost in Eukaryotes.[25] Its N-terminal HSH domain is similar to AAA proteins, especially Pol III subunit δ and RuvB, in structure.[39] DP2 has a Class II KH domain.[17] Pyrococcus abyssi polD is more heat-stable and more accurate than Taq polymerase, but has not yet been commercialized.[40] It has been proposed that family D DNA polymerase was the first to evolve in cellular organisms and that the replicative polymerase of the Last Universal Cellular Ancestor (LUCA) belonged to family D.[41]
Eukaryotic DNA polymerase[edit]
Polymerases β, λ, σ, μ (beta, lambda, sigma, mu) and TdT[edit]
Family X polymerases contain the well-known eukaryotic polymerase pol β (beta), as well as other eukaryotic polymerases such as Pol σ (sigma), Pol λ (lambda), Pol μ (mu), and Terminal deoxynucleotidyl transferase (TdT). Family X polymerases are found mainly in vertebrates, and a few are found in plants and fungi. These polymerases have highly conserved regions that include two helix-hairpin-helix motifs that are imperative in the DNA-polymerase interactions. One motif is located in the 8 kDa domain that interacts with downstream DNA and one motif is located in the thumb domain that interacts with the primer strand. Pol β, encoded by POLB gene, is required for short-patch base excision repair, a DNA repair pathway that is essential for repairing alkylated or oxidized bases as well as abasic sites. Pol λ and Pol μ, encoded by the POLL and POLM genes respectively, are involved in non-homologous end-joining, a mechanism for rejoining DNA double-strand breaks due to hydrogen peroxide and ionizing radiation, respectively. TdT is expressed only in lymphoid tissue, and adds «n nucleotides» to double-strand breaks formed during V(D)J recombination to promote immunological diversity.[42]
Polymerases α, δ and ε (alpha, delta, and epsilon)[edit]
Pol α (alpha), Pol δ (delta), and Pol ε (epsilon) are members of Family B Polymerases and are the main polymerases involved with nuclear DNA replication. Pol α complex (pol α-DNA primase complex) consists of four subunits: the catalytic subunit POLA1, the regulatory subunit POLA2, and the small and the large primase subunits PRIM1 and PRIM2 respectively. Once primase has created the RNA primer, Pol α starts replication elongating the primer with ~20 nucleotides.[43] Due to its high processivity, Pol δ takes over the leading and lagging strand synthesis from Pol α.[14]: 218–219 Pol δ is expressed by genes POLD1, creating the catalytic subunit, POLD2, POLD3, and POLD4 creating the other subunits that interact with Proliferating Cell Nuclear Antigen (PCNA), which is a DNA clamp that allows Pol δ to possess processivity.[44] Pol ε is encoded by the POLE1, the catalytic subunit, POLE2, and POLE3 gene. It has been reported that the function of Pol ε is to extend the leading strand during replication,[45][46] while Pol δ primarily replicates the lagging strand; however, recent evidence suggested that Pol δ might have a role in replicating the leading strand of DNA as well.[47] Pol ε’s C-terminus «polymerase relic» region, despite being unnecessary for polymerase activity,[48] is thought to be essential to cell vitality. The C-terminus region is thought to provide a checkpoint before entering anaphase, provide stability to the holoenzyme, and add proteins to the holoenzyme necessary for initiation of replication.[49] Pol ε has a larger «palm» domain that provides high processivity independently of PCNA.[48]
Compared to other Family B polymerases, the DEDD exonuclease family responsible for proofreading is inactivated in Pol α.[25] Pol ε is unique in that it has two zinc finger domains and an inactive copy of another family B polymerase in its C-terminal. The presence of this zinc finger has implications in the origins of Eukaryota, which in this case is placed into the Asgard group with archaeal B3 polymerase.[50]
Polymerases η, ι and κ (eta, iota, and kappa)[edit]
Pol η (eta), Pol ι (iota), and Pol κ (kappa), are Family Y DNA polymerases involved in the DNA repair by translation synthesis and encoded by genes POLH, POLI, and POLK respectively. Members of Family Y have five common motifs to aid in binding the substrate and primer terminus and they all include the typical right hand thumb, palm and finger domains with added domains like little finger (LF), polymerase-associated domain (PAD), or wrist. The active site, however, differs between family members due to the different lesions being repaired. Polymerases in Family Y are low-fidelity polymerases, but have been proven to do more good than harm as mutations that affect the polymerase can cause various diseases, such as skin cancer and Xeroderma Pigmentosum Variant (XPS). The importance of these polymerases is evidenced by the fact that gene encoding DNA polymerase η is referred as XPV, because loss of this gene results in the disease Xeroderma Pigmentosum Variant. Pol η is particularly important for allowing accurate translesion synthesis of DNA damage resulting from ultraviolet radiation. The functionality of Pol κ is not completely understood, but researchers have found two probable functions. Pol κ is thought to act as an extender or an inserter of a specific base at certain DNA lesions. All three translesion synthesis polymerases, along with Rev1, are recruited to damaged lesions via stalled replicative DNA polymerases. There are two pathways of damage repair leading researchers to conclude that the chosen pathway depends on which strand contains the damage, the leading or lagging strand.[51]
Polymerases Rev1 and ζ (zeta)[edit]
Pol ζ another B family polymerase, is made of two subunits Rev3, the catalytic subunit, and Rev7 (MAD2L2), which increases the catalytic function of the polymerase, and is involved in translesion synthesis. Pol ζ lacks 3′ to 5′ exonuclease activity, is unique in that it can extend primers with terminal mismatches. Rev1 has three regions of interest in the BRCT domain, ubiquitin-binding domain, and C-terminal domain and has dCMP transferase ability, which adds deoxycytidine opposite lesions that would stall replicative polymerases Pol δ and Pol ε. These stalled polymerases activate ubiquitin complexes that in turn disassociate replication polymerases and recruit Pol ζ and Rev1. Together Pol ζ and Rev1 add deoxycytidine and Pol ζ extends past the lesion. Through a yet undetermined process, Pol ζ disassociates and replication polymerases reassociate and continue replication. Pol ζ and Rev1 are not required for replication, but loss of REV3 gene in budding yeast can cause increased sensitivity to DNA-damaging agents due to collapse of replication forks where replication polymerases have stalled.[52]
Telomerase[edit]
Telomerase is a ribonucleoprotein which functions to replicate ends of linear chromosomes since normal DNA polymerase cannot replicate the ends, or telomeres. The single-strand 3′ overhang of the double-strand chromosome with the sequence 5′-TTAGGG-3′ recruits telomerase. Telomerase acts like other DNA polymerases by extending the 3′ end, but, unlike other DNA polymerases, telomerase does not require a template. The TERT subunit, an example of a reverse transcriptase, uses the RNA subunit to form the primer–template junction that allows telomerase to extend the 3′ end of chromosome ends. The gradual decrease in size of telomeres as the result of many replications over a lifetime are thought to be associated with the effects of aging.[14]: 248–249
Polymerases γ, θ and ν (gamma, theta and nu)[edit]
Pol γ (gamma), Pol θ (theta), and Pol ν (nu) are Family A polymerases. Pol γ, encoded by the POLG gene, was long thought to be the only mitochondrial polymerase. However, recent research shows that at least Pol β (beta), a Family X polymerase, is also present in mitochondria.[53][54] Any mutation that leads to limited or non-functioning Pol γ has a significant effect on mtDNA and is the most common cause of autosomal inherited mitochondrial disorders.[55] Pol γ contains a C-terminus polymerase domain and an N-terminus 3’–5′ exonuclease domain that are connected via the linker region, which binds the accessory subunit. The accessory subunit binds DNA and is required for processivity of Pol γ. Point mutation A467T in the linker region is responsible for more than one-third of all Pol γ-associated mitochondrial disorders.[56] While many homologs of Pol θ, encoded by the POLQ gene, are found in eukaryotes, its function is not clearly understood. The sequence of amino acids in the C-terminus is what classifies Pol θ as Family A polymerase, although the error rate for Pol θ is more closely related to Family Y polymerases. Pol θ extends mismatched primer termini and can bypass abasic sites by adding a nucleotide. It also has Deoxyribophosphodiesterase (dRPase) activity in the polymerase domain and can show ATPase activity in close proximity to ssDNA.[57] Pol ν (nu) is considered to be the least effective of the polymerase enzymes.[58] However, DNA polymerase nu plays an active role in homology repair during cellular responses to crosslinks, fulfilling its role in a complex with helicase.[58]
Plants use two Family A polymerases to copy both the mitochondrial and plastid genomes. They are more similar to bacterial Pol I than they are to mammalian Pol γ.[59]
Reverse transcriptase[edit]
Retroviruses encode an unusual DNA polymerase called reverse transcriptase, which is an RNA-dependent DNA polymerase (RdDp) that synthesizes DNA from a template of RNA. The reverse transcriptase family contain both DNA polymerase functionality and RNase H functionality, which degrades RNA base-paired to DNA. An example of a retrovirus is HIV.[14] Reverse transcriptase is commonly employed in amplification of RNA for research purposes. Using an RNA template, PCR can utilize reverse transcriptase, creating a DNA template. This new DNA template can then be used for typical PCR amplification. The products of such an experiment are thus amplified PCR products from RNA.[9]
Each HIV retrovirus particle contains two RNA genomes, but, after an infection, each virus generates only one provirus.[60] After infection, reverse transcription is accompanied by template switching between the two genome copies (copy choice recombination).[60] From 5 to 14 recombination events per genome occur at each replication cycle.[61] Template switching (recombination) appears to be necessary for maintaining genome integrity and as a repair mechanism for salvaging damaged genomes.[62][60]
Bacteriophage T4 DNA polymerase[edit]
Bacteriophage (phage) T4 encodes a DNA polymerase that catalyzes DNA synthesis in a 5′ to 3′ direction.[63] The phage polymerase also has an exonuclease activity that acts in a 3′ to 5′ direction,[64] and this activity is employed in the proofreading and editing of newly inserted bases.[65] A phage mutant with a temperature sensitive DNA polymerase, when grown at permissive temperatures, was observed to undergo recombination at frequencies that are about two-fold higher than that of wild-type phage.[66]
It was proposed that a mutational alteration in the phage DNA polymerase can stimulate template strand switching (copy choice recombination) during replication.[66]
See also[edit]
- Biological machines
- DNA sequencing
- Enzyme catalysis
- Genetic recombination
- Molecular cloning
- Polymerase chain reaction
- Protein domain dynamics
- Reverse transcription
- RNA polymerase
- Taq DNA polymerase
References[edit]
- ^ Bollum FJ (August 1960). «Calf thymus polymerase». The Journal of Biological Chemistry. 235 (8): 2399–403. doi:10.1016/S0021-9258(18)64634-4. PMID 13802334.
- ^ Falaschi A, Kornberg A (April 1966). «Biochemical studies of bacterial sporulation. II. Deoxy- ribonucleic acid polymerase in spores of Bacillus subtilis». The Journal of Biological Chemistry. 241 (7): 1478–82. doi:10.1016/S0021-9258(18)96736-0. PMID 4957767.
- ^ Lehman IR, Bessman MJ, Simms ES, Kornberg A (July 1958). «Enzymatic synthesis of deoxyribonucleic acid. I. Preparation of substrates and partial purification of an enzyme from Escherichia coli». The Journal of Biological Chemistry. 233 (1): 163–70. doi:10.1016/S0021-9258(19)68048-8. PMID 13563462.
- ^ Richardson CC, Schildkraut CL, Aposhian HV, Kornberg A (January 1964). «Enzymatic synthesis of deoxyribonucleic acid. XIV. Further purification and properties of deoxyribonucleic acid polymerase of Escherichia coli«. The Journal of Biological Chemistry. 239: 222–32. doi:10.1016/S0021-9258(18)51772-5. PMID 14114848.
- ^ Schachman HK, Adler J, Radding CM, Lehman IR, Kornberg A (November 1960). «Enzymatic synthesis of deoxyribonucleic acid. VII. Synthesis of a polymer of deoxyadenylate and deoxythymidylate». The Journal of Biological Chemistry. 235 (11): 3242–9. doi:10.1016/S0021-9258(20)81345-3. PMID 13747134.
- ^ Zimmerman BK (May 1966). «Purification and properties of deoxyribonucleic acid polymerase from Micrococcus lysodeikticus». The Journal of Biological Chemistry. 241 (9): 2035–41. doi:10.1016/S0021-9258(18)96662-7. PMID 5946628.
- ^ «The Nobel Prize in Physiology or Medicine 1959». Nobel Foundation. Retrieved December 1, 2012.
- ^ Tessman I, Kennedy MA (February 1994). «DNA polymerase II of Escherichia coli in the bypass of abasic sites in vivo». Genetics. 136 (2): 439–48. doi:10.1093/genetics/136.2.439. PMC 1205799. PMID 7908652.
- ^ a b Lehninger AL, Cox MM, Nelson DL (2013). Lehninger principles of biochemistry (6th ed.). New York: W.H. Freeman and Company. ISBN 978-1-4292-3414-6. OCLC 824794893.
- ^ Garrett G (2013). Biochemistry. Mary Finch.
- ^ Hunter WN, Brown T, Anand NN, Kennard O (1986). «Structure of an adenine-cytosine base pair in DNA and its implications for mismatch repair». Nature. 320 (6062): 552–5. Bibcode:1986Natur.320..552H. doi:10.1038/320552a0. PMID 3960137. S2CID 4319887.
- ^ Swan MK, Johnson RE, Prakash L, Prakash S, Aggarwal AK (September 2009). «Structural basis of high-fidelity DNA synthesis by yeast DNA polymerase delta». Nature Structural & Molecular Biology. 16 (9): 979–86. doi:10.1038/nsmb.1663. PMC 3055789. PMID 19718023.
- ^ Steitz TA (June 1999). «DNA polymerases: structural diversity and common mechanisms». The Journal of Biological Chemistry. 274 (25): 17395–8. doi:10.1074/jbc.274.25.17395. PMID 10364165.
- ^ a b c d e Losick R, Watson JD, Baker TA, Bell S, Gann A, Levine MW (2008). Molecular biology of the gene (6th ed.). San Francisco: Pearson/Benjamin Cummings. ISBN 978-0-8053-9592-1.
- ^ McCarthy D, Minner C, Bernstein H, Bernstein C (October 1976). «DNA elongation rates and growing point distributions of wild-type phage T4 and a DNA-delay amber mutant». Journal of Molecular Biology. 106 (4): 963–81. doi:10.1016/0022-2836(76)90346-6. PMID 789903.
- ^ Filée J, Forterre P, Sen-Lin T, Laurent J (June 2002). «Evolution of DNA polymerase families: evidences for multiple gene exchange between cellular and viral proteins» (PDF). Journal of Molecular Evolution. 54 (6): 763–73. Bibcode:2002JMolE..54..763F. CiteSeerX 10.1.1.327.4738. doi:10.1007/s00239-001-0078-x. PMID 12029358. S2CID 15852365. Archived from the original (PDF) on 2020-07-29. Retrieved 2019-09-23.
- ^ a b c Raia P, Carroni M, Henry E, Pehau-Arnaudet G, Brûlé S, Béguin P, Henneke G, Lindahl E, Delarue M, Sauguet L (January 2019). «Structure of the DP1-DP2 PolD complex bound with DNA and its implications for the evolutionary history of DNA and RNA polymerases». PLOS Biology. 17 (1): e3000122. doi:10.1371/journal.pbio.3000122. PMC 6355029. PMID 30657780.
- ^ Boehm EM, Powers KT, Kondratick CM, Spies M, Houtman JC, Washington MT (April 2016). «The Proliferating Cell Nuclear Antigen (PCNA)-interacting Protein (PIP) Motif of DNA Polymerase η Mediates Its Interaction with the C-terminal Domain of Rev1». The Journal of Biological Chemistry. 291 (16): 8735–44. doi:10.1074/jbc.M115.697938. PMC 4861442. PMID 26903512.
- ^ Yang W (May 2014). «An overview of Y-Family DNA polymerases and a case study of human DNA polymerase η». Biochemistry. 53 (17): 2793–803. doi:10.1021/bi500019s. PMC 4018060. PMID 24716551.
- ^ Maga G, Hubscher U, Spadari S, Villani G (2010). DNA Polymerases: Discovery, Characterization Functions in Cellular DNA Transactions. World Scientific Publishing Company. ISBN 978-981-4299-16-9.
- ^ Choi CH, Burton ZF, Usheva A (February 2004). «Auto-acetylation of transcription factors as a control mechanism in gene expression». Cell Cycle. 3 (2): 114–5. doi:10.4161/cc.3.2.651. PMID 14712067.
- ^ Chien A, Edgar DB, Trela JM (September 1976). «Deoxyribonucleic acid polymerase from the extreme thermophile Thermus aquaticus». Journal of Bacteriology. 127 (3): 1550–7. doi:10.1128/JB.127.3.1550-1557.1976. PMC 232952. PMID 8432.
- ^ a b Banach-Orlowska M, Fijalkowska IJ, Schaaper RM, Jonczyk P (October 2005). «DNA polymerase II as a fidelity factor in chromosomal DNA synthesis in Escherichia coli». Molecular Microbiology. 58 (1): 61–70. doi:10.1111/j.1365-2958.2005.04805.x. PMID 16164549. S2CID 12002486.
- ^ InterPro protein view: P61875
- ^ a b c Makarova KS, Krupovic M, Koonin EV (2014). «Evolution of replicative DNA polymerases in archaea and their contributions to the eukaryotic replication machinery». Frontiers in Microbiology. 5: 354. doi:10.3389/fmicb.2014.00354. PMC 4104785. PMID 25101062.
- ^ Rohe M, Schrage K, Meinhardt F (December 1991). «The linear plasmid pMC3-2 from Morchella conica is structurally related to adenoviruses». Current Genetics. 20 (6): 527–33. doi:10.1007/BF00334782. PMID 1782679. S2CID 35072924.
- ^ Olson MW, Dallmann HG, McHenry CS (December 1995). «DnaX complex of Escherichia coli DNA polymerase III holoenzyme. The chi psi complex functions by increasing the affinity of tau and gamma for delta.delta’ to a physiologically relevant range». The Journal of Biological Chemistry. 270 (49): 29570–7. doi:10.1074/jbc.270.49.29570. PMID 7494000.
- ^ Liao Y, Li Y, Schroeder JW, Simmons LA, Biteen JS (December 2016). «Single-Molecule DNA Polymerase Dynamics at a Bacterial Replisome in Live Cells». Biophysical Journal. 111 (12): 2562–2569. Bibcode:2016BpJ…111.2562L. doi:10.1016/j.bpj.2016.11.006. PMC 5192695. PMID 28002733.
- ^ a b c Xu ZQ, Dixon NE (December 2018). «Bacterial replisomes». Current Opinion in Structural Biology. 53: 159–168. doi:10.1016/j.sbi.2018.09.006. PMID 30292863.
- ^ Goodman MF (2002). «Error-prone repair DNA polymerases in prokaryotes and eukaryotes». Annual Review of Biochemistry. 71: 17–50. doi:10.1146/annurev.biochem.71.083101.124707. PMID 12045089. S2CID 1979429.
- ^ Mori T, Nakamura T, Okazaki N, Furukohri A, Maki H, Akiyama MT (2012). «Escherichia coli DinB inhibits replication fork progression without significantly inducing the SOS response». Genes & Genetic Systems. 87 (2): 75–87. doi:10.1266/ggs.87.75. PMID 22820381.
- ^ Jarosz DF, Godoy VG, Walker GC (April 2007). «Proficient and accurate bypass of persistent DNA lesions by DinB DNA polymerases». Cell Cycle. 6 (7): 817–22. doi:10.4161/cc.6.7.4065. PMID 17377496.
- ^ Patel M, Jiang Q, Woodgate R, Cox MM, Goodman MF (June 2010). «A new model for SOS-induced mutagenesis: how RecA protein activates DNA polymerase V». Critical Reviews in Biochemistry and Molecular Biology. 45 (3): 171–84. doi:10.3109/10409238.2010.480968. PMC 2874081. PMID 20441441.
- ^ Sutton MD, Walker GC (July 2001). «Managing DNA polymerases: coordinating DNA replication, DNA repair, and DNA recombination». Proceedings of the National Academy of Sciences of the United States of America. 98 (15): 8342–9. Bibcode:2001PNAS…98.8342S. doi:10.1073/pnas.111036998. PMC 37441. PMID 11459973.
- ^ a b Raychaudhury P, Basu AK (March 2011). «Genetic requirement for mutagenesis of the G[8,5-Me]T cross-link in Escherichia coli: DNA polymerases IV and V compete for error-prone bypass». Biochemistry. 50 (12): 2330–8. doi:10.1021/bi102064z. PMC 3062377. PMID 21302943.
- ^ Madru C, Henneke G, Raia P, Hugonneau-Beaufet I, Pehau-Arnaudet G, England P, et al. (March 2020). «Structural basis for the increased processivity of D-family DNA polymerases in complex with PCNA». Nature Communications. 11 (1): 1591. doi:10.1038/s41467-020-15392-9. PMID 32221299.
- ^ Ishino Y, Komori K, Cann IK, Koga Y (April 1998). «A novel DNA polymerase family found in Archaea». Journal of Bacteriology. 180 (8): 2232–6. doi:10.1128/JB.180.8.2232-2236.1998. PMC 107154. PMID 9555910.
- ^ Sauguet L, Raia P, Henneke G, Delarue M (2016). «Shared active site architecture between archaeal PolD and multi-subunit RNA polymerases revealed by X-ray crystallography». Nature Communications. 7: 12227. Bibcode:2016NatCo…712227S. doi:10.1038/ncomms12227. PMC 4996933. PMID 27548043.
- ^ Yamasaki K, Urushibata Y, Yamasaki T, Arisaka F, Matsui I (August 2010). «Solution structure of the N-terminal domain of the archaeal D-family DNA polymerase small subunit reveals evolutionary relationship to eukaryotic B-family polymerases». FEBS Letters. 584 (15): 3370–5. doi:10.1016/j.febslet.2010.06.026. PMID 20598295. S2CID 11229530.
- ^ Ishino S, Ishino Y (2014). «DNA polymerases as useful reagents for biotechnology — the history of developmental research in the field». Frontiers in Microbiology. 5: 465. doi:10.3389/fmicb.2014.00465. PMC 4148896. PMID 25221550.
- ^ Koonin EV, Krupovic M, Ishino S, Ishino Y (June 2020). «The replication machinery of LUCA: common origin of DNA replication and transcription». BMC Biology. 18 (1): 61. doi:10.1186/s12915-020-00800-9. PMC 7281927. PMID 32517760.
- ^ Yamtich J, Sweasy JB (May 2010). «DNA polymerase family X: function, structure, and cellular roles». Biochimica et Biophysica Acta (BBA) — Proteins and Proteomics. 1804 (5): 1136–50. doi:10.1016/j.bbapap.2009.07.008. PMC 2846199. PMID 19631767.
- ^ Chansky ML, Marks A, Peet A (2012). Marks’ Basic Medical Biochemistry: a clinical approach (4th ed.). Philadelphia: Wolter Kluwer Health/Lippincott Williams & Wilkins. p. chapter13. ISBN 978-1608315727.
- ^ Chung DW, Zhang JA, Tan CK, Davie EW, So AG, Downey KM (December 1991). «Primary structure of the catalytic subunit of human DNA polymerase delta and chromosomal location of the gene». Proceedings of the National Academy of Sciences of the United States of America. 88 (24): 11197–201. Bibcode:1991PNAS…8811197C. doi:10.1073/pnas.88.24.11197. PMC 53101. PMID 1722322.
- ^ Pursell ZF, Isoz I, Lundström EB, Johansson E, Kunkel TA (July 2007). «Yeast DNA polymerase epsilon participates in leading-strand DNA replication». Science. 317 (5834): 127–30. Bibcode:2007Sci…317..127P. doi:10.1126/science.1144067. PMC 2233713. PMID 17615360.
- ^ Lujan SA, Williams JS, Kunkel TA (September 2016). «DNA Polymerases Divide the Labor of Genome Replication». Trends in Cell Biology. 26 (9): 640–654. doi:10.1016/j.tcb.2016.04.012. PMC 4993630. PMID 27262731.
- ^ Johnson RE, Klassen R, Prakash L, Prakash S (July 2015). «A Major Role of DNA Polymerase δ in Replication of Both the Leading and Lagging DNA Strands». Molecular Cell. 59 (2): 163–175. doi:10.1016/j.molcel.2015.05.038. PMC 4517859. PMID 26145172.
- ^ a b Doublié S, Zahn KE (2014). «Structural insights into eukaryotic DNA replication». Frontiers in Microbiology. 5: 444. doi:10.3389/fmicb.2014.00444. PMC 4142720. PMID 25202305.
- ^ Edwards S, Li CM, Levy DL, Brown J, Snow PM, Campbell JL (April 2003). «Saccharomyces cerevisiae DNA polymerase epsilon and polymerase sigma interact physically and functionally, suggesting a role for polymerase epsilon in sister chromatid cohesion». Molecular and Cellular Biology. 23 (8): 2733–48. doi:10.1128/mcb.23.8.2733-2748.2003. PMC 152548. PMID 12665575.
- ^ Zaremba-Niedzwiedzka K, Caceres EF, Saw JH, Bäckström D, Juzokaite L, Vancaester E, Seitz KW, Anantharaman K, Starnawski P, Kjeldsen KU, Stott MB, Nunoura T, Banfield JF, Schramm A, Baker BJ, Spang A, Ettema TJ (January 2017). «Asgard archaea illuminate the origin of eukaryotic cellular complexity». Nature. 541 (7637): 353–358. Bibcode:2017Natur.541..353Z. doi:10.1038/nature21031. OSTI 1580084. PMID 28077874. S2CID 4458094.
- ^ Ohmori H, Hanafusa T, Ohashi E, Vaziri C (2009). Separate roles of structured and unstructured regions of Y-family DNA polymerases. Advances in Protein Chemistry and Structural Biology. Vol. 78. pp. 99–146. doi:10.1016/S1876-1623(08)78004-0. ISBN 9780123748270. PMC 3103052. PMID 20663485.
- ^ Gan GN, Wittschieben JP, Wittschieben BØ, Wood RD (January 2008). «DNA polymerase zeta (pol zeta) in higher eukaryotes». Cell Research. 18 (1): 174–83. doi:10.1038/cr.2007.117. PMID 18157155.
- ^ Bienstock R, Beard W, Wilson S (August 2014). «Phylogenetic analysis and evolutionary origins of DNA polymerase X-family members». DNA Repair. 22: 77–88. doi:10.1016/j.dnarep.2014.07.003. PMC 4260717. PMID 25112931.
- ^ Prasad R, et al. (October 2017). «DNA polymerase β: A missing link of the base excision repair machinery in mammalian mitochondria». DNA Repair. 60: 77–88. doi:10.1016/j.dnarep.2017.10.011. PMC 5919216. PMID 29100041.
- ^ Zhang L, Chan SS, Wolff DJ (July 2011). «Mitochondrial disorders of DNA polymerase γ dysfunction: from anatomic to molecular pathology diagnosis». Archives of Pathology & Laboratory Medicine. 135 (7): 925–34. doi:10.5858/2010-0356-RAR.1. PMC 3158670. PMID 21732785.
- ^ Stumpf JD, Copeland WC (January 2011). «Mitochondrial DNA replication and disease: insights from DNA polymerase γ mutations». Cellular and Molecular Life Sciences. 68 (2): 219–33. doi:10.1007/s00018-010-0530-4. PMC 3046768. PMID 20927567.
- ^ Hogg M, Sauer-Eriksson AE, Johansson E (March 2012). «Promiscuous DNA synthesis by human DNA polymerase θ». Nucleic Acids Research. 40 (6): 2611–22. doi:10.1093/nar/gkr1102. PMC 3315306. PMID 22135286.
- ^ a b «UniProtKB — Q7Z5Q5 (DPOLN_HUMAN)». Uniprot. Retrieved 5 July 2018.
- ^ Cupp JD, Nielsen BL (November 2014). «Minireview: DNA replication in plant mitochondria». Mitochondrion. 19 Pt B: 231–7. doi:10.1016/j.mito.2014.03.008. PMC 417701. PMID 24681310.
- ^ a b c Rawson JM, Nikolaitchik OA, Keele BF, Pathak VK, Hu WS (November 2018). «Recombination is required for efficient HIV-1 replication and the maintenance of viral genome integrity». Nucleic Acids Research. 46 (20): 10535–10545. doi:10.1093/nar/gky910. PMC 6237782. PMID 30307534.
- ^ Cromer D, Grimm AJ, Schlub TE, Mak J, Davenport MP (January 2016). «Estimating the in-vivo HIV template switching and recombination rate». AIDS. 30 (2): 185–92. doi:10.1097/QAD.0000000000000936. PMID 26691546. S2CID 20086739.
- ^ Hu WS, Temin HM (November 1990). «Retroviral recombination and reverse transcription». Science. 250 (4985): 1227–33. Bibcode:1990Sci…250.1227H. doi:10.1126/science.1700865. PMID 1700865.
- ^ Goulian M, Lucas ZJ, Kornberg A (February 1968). «Enzymatic synthesis of deoxyribonucleic acid. XXV. Purification and properties of deoxyribonucleic acid polymerase induced by infection with phage T4». The Journal of Biological Chemistry. 243 (3): 627–38. doi:10.1016/S0021-9258(18)93650-1. PMID 4866523.
- ^ Huang WM, Lehman IR (May 1972). «On the exonuclease activity of phage T4 deoxyribonucleic acid polymerase». The Journal of Biological Chemistry. 247 (10): 3139–46. doi:10.1016/S0021-9258(19)45224-1. PMID 4554914.
- ^ Gillin FD, Nossal NG (September 1976). «Control of mutation frequency by bacteriophage T4 DNA polymerase. I. The CB120 antimutator DNA polymerase is defective in strand displacement». The Journal of Biological Chemistry. 251 (17): 5219–24. doi:10.1016/S0021-9258(17)33149-6. PMID 956182.
- ^ a b Bernstein H (August 1967). «The effect on recombination of mutational defects in the DNA-polymerase and deoxycytidylate hydroxymethylase of phage T4D». Genetics. 56 (4): 755–69. doi:10.1093/genetics/56.4.755. PMC 1211652. PMID 6061665.
Further reading[edit]
- Burgers PM, Koonin EV, Bruford E, Blanco L, Burtis KC, Christman MF, Copeland WC, Friedberg EC, Hanaoka F, Hinkle DC, Lawrence CW, Nakanishi M, Ohmori H, Prakash L, Prakash S, Reynaud CA, Sugino A, Todo T, Wang Z, Weill JC, Woodgate R (November 2001). «Eukaryotic DNA polymerases: proposal for a revised nomenclature». The Journal of Biological Chemistry. 276 (47): 43487–90. doi:10.1074/jbc.R100056200. PMID 11579108.
External links[edit]
- DNA+polymerases at the US National Library of Medicine Medical Subject Headings (MeSH)
- PDB Molecule of the Month DNA polymerase
- Unusual repair mechanism in DNA polymerase lambda, Ohio State University, July 25, 2006.
- A great animation of DNA Polymerase from WEHI at 1:45 minutes in
- 3D macromolecular structures of DNA polymerase from the EM Data Bank(EMDB)
- Авторы
- Резюме
- Файлы
- Ключевые слова
- Литература
Лопатина А.Б.
1
1 ГОУ ВПО «Пермский национальный исследовательский политехнический университет»
В данном научном обзоре рассматриваются теоретические химического обеспечения механизмов репарации ДНК микробиологических систем на примере одноклеточных структур – бактерий. Описаны понятия самовосстановления и репарации ДНК за счет функционирования ферментных систем. Описаны механизмы распознавания повреждений, систем узнавания и выявления поврежденных участков и процессы подключения способов устранения поломок. Большое внимание уделяется механизмам повреждения бактерий ультрафиолетом. Однако описывается и механизм восстановления ДНК с помощью солнечного света. Подробно освещены механизмы репарации ДНК, такие как: фотореактивация, мисматч-репарация, эксцизионная репарация нуклеотидов, рекомбинационная репарация, воссоединение негомологичных концов. Все эти механизмы, изучаемые на примере одноклеточных структур, имеют место быть и в структурах многоклеточных, включая и организм человека. Поэтому изучение химического и ферментного обеспечения механизмов репарации ДНК на примере микробиологических систем является важной и актуальной задачей современной химии.
ДНК
репарация
фотореактивация
ферменты
1. Кочетков Н.К., Будовский Э.И., Свердлов Е.Д., Симукова Н.А., Турчинский М.Ф., Шибаев В.Н. Органическая химия нуклеиновых кислот. – М., Химия, 1970. – 718 с.
2. Сетлоу Р., Поллард Э. Молекулярная биофизика. –М.: Мир, 1964. – 440 с.
3. Dulbecco R., and Freeman, G., Virology, 8, 396 (1959).
4. Lindahl T. New class of enzymes acting on damaged DNA // Nature. – 1976. – V. 259. – P. 64–66.
5. Lindahl T. Instability and decay of the primary structure of DNA // Nature. – 1993. – V. 362. P. 709–715.
6. Modrich P. Methyl-directed repair of DNA base pair mismatches in vitro // Proc. Natl Acad. Sci. USA. – 1983. – V. 80. – P. 4639–4643.
7 Paul Modrich P. Mechanisms and biological effects of mismatch repair // Annu. Rev. Genet. – 1991. – V. 25. – P. 229–253.
8. Rupert C.S., Goodgal Sol H., and Herriott, Roger M., 1958, Photoreactivation in vitro of Ultraviolet Inactivated Hemophilus in fluenzae Transforming Factor, Journal of General Physiology, 41: 451–471.
9. Sancar A. A novel repair enzyme: UVRABC excision nuclease of Escherichia coli cuts a DNA strand on both sides of the damaged region // Cell. – 1983. – V. 33. – P. 249–260.
10. Sancar A. Structure and function of DNA photolyase // Biochemistry. – 1994. – V. 33. – P. 2–22.
11. Setlow, Richard B., 1997, DNA Damage and Repair: A Photobiological Odyssey, Photochemistry and Photobiology, 65S: 119S-122S.
В настоящее время современная наука все больше и больше внимания уделяет нанообъектам, молекулярной биологии и молекулярной химии. Безусловно, что первыми объектами для изучения на молекулярном уровне становятся одноклеточные структуры, такие как бактерии, вирусы. ДНК этих структур, являющихся носителями информации, в первую очередь подвергается расщеплению, попыткам синтеза, подвергается различным облучениям, солнечному, радиоактивному, с целью исследования ответной реакции на воздействие этих факторов. Безусловно, изучение химических реакций на уровне микробиологических структур экстраполируется на попытки понимания течения подобных процессов и реакций в структурах живого человеческого организма, что и делает эти опыты актуальными и важными. Исследование закономерности мутационных процессов на уровне клеток, а затем и организма человека в целом невозможно без понимания закономерностей происхождения мутаций на уровне одноклеточных субъектов. Попытки изучения ДНК и химических реакций, протекающих в клетках бактерий насчитывают уже десятки лет. Успехи современной химии, биохимии и молекулярной химии являются огромными, но даже при таком прогрессивном изучении молекулярных и субмолекулярных процессов загадки природы, сотворившей и встроившей процессы самовосстановления, саморегуляции и репарации ДНК, превосходят все мыслимые и немыслимые ожидания. 2015 год ознаменовался вручением Нобелевской премии трем ученым за вклад в изучение механизмов репарации ДНК. Целью данной работы является описание теоретических основ химического обеспечения механизмов репарации ДНК микробиологических систем, с экстраполяцией полученных результатов на макроуровень, в том числе на уровень организма человека, что делает изучение этой проблемы важной и актуальной задачей.
Исследования ДНК на одноклеточных организмах, бактериях выявили механизм восстановления ДНК бактерий после повреждений, нанесенных ультрафиолетом, при воздействии этого же самого солнечного света. То есть тот фактор, который явялется повреждающим, является также и восстанавливающим. Это явление было названо фотореактивацией и положило основу для дальнейших исследований механизмов репарации ДНК [9]. Подобные результаты исследований получили и другие ученые – Альберт Кельнер и Нобелевский лауреат, вирусолог [3].
В настоящее время известно несколько разных механизмов репарации ДНК. ДНК всех живых организмов постоянно подвергается воздействию повреждающих факторов: ультрафиолет, радиация, тысячи химически активных веществ в нашей пище, химические соединения, содержащиеся в кофе и кофейных напитках. Но гораздо важнее факторы внутренние, которых мы не можем избежать в принципе. Главных таких факторов три. Во-первых, весь наш обмен веществ основан на кислородном дыхании. Митохондрии – клеточные органеллы, в которых кислород используется для производства АТФ, «энергетической валюты» наших клеток, – работают не с абсолютной эффективностью, и промежуточные активные формы кислорода утекают из них и способны повреждать ДНК. Во-вторых, как известно, мы в среднем на 60 % состоим из воды, которая, в общем, тоже очень активное соединение и постоянно гидролизует ДНК. Наконец, еще одним важным источником повреждений в ДНК служат ошибки ферментов, которые ее копируют, – ДНК-полимераз; количество неверно включенных нуклеотидов составляет около 300 000 на каждое клеточное деление.
Фотореактивации – один из частных примеров механизма реактивации, или прямого восстановления, при котором поврежденное звено ДНК превращается в нормальное без каких-то промежуточных шагов. В случае фотореактивации происходит вот что. Под влиянием ультрафиолетового света соседние основания тимина в ДНК могут сшиваться друг с другом и образовывать так называемые циклобутановые пиримидиновые димеры, которые очень сильно искажают структуру ДНК и не дают возможности ДНК-полимеразам копировать поврежденный участок. Бактерии же содержат фермент фотолиазу, который использует энергию видимого света для того, чтобы расщепить связи между основаниями в димере, превращая его опять в два тимина.
Фотолиазу открыл в конце 1950-х годов Стэн Руперт (Stan Rupert) [7], с которым когда-то работал нынешний нобелевский лауреат Азиз Санджар, который впервые клонировал фотолиазу, то есть выделил кодирующий ее ген, а потом произвел генно-инженерный белок. Тем самым Санджар сумел произвести изучаемый белок в нужных для исследования количествах, поскольку природной фотолиазы в бактериях очень мало. Фотолиаза – это пример сложной химической системы, осуществляющей фотокатализ: путь энергии, принесенной фотоном, поглощенным 5,10-метенилтетрагидроптероилполиглутаматом – хромофором в составе белка – через второй хромофор (флавинадениндинуклеотид) к циклобутановому пиримидиновому димеру сейчас прослежен вплоть до квантовомеханического описания. Помимо этого Санджар изучал и явление «темновой репарации». Бактерии, облученные ультрафиолетом, способны исправлять внесенные повреждения не только на свету – просто для этого нужно гораздо больше времени. Фотолиаза помогает темновой репарации, но без нее вполне можно обойтись, так как в эту работу включаются другие ферменты.
К тому времени было известно, что в темноте тиминовые димеры постепенно исчезают из ДНК (это открытие сделал в начале 1960-х годов Ричард Сетлоу (Richard B. Setlow) [2, 10]. После облучения ультрафиолетом в клетках начинается синтез ДНК (автор этого открытия Филип Ханаволт (Philip Hanawalt). Были известны три гена, которые отвечали за темновую репарацию, их назвали uvrA, uvrB и uvrC (uvr – от английского «UV-resistant», устойчивый к ультрафиолету), но оставалось совершенно непонятно, как же всё это в клетке происходит. Опять же, в основном проблемы были в том, что белков этих в клетке очень мало, и исследовать их из-за этого очень трудно.
Санджар изобрел метод бактериальных «макси-клеток», который позволял получать огромный избыток нужного продукта при минимальном загрязнении другими клеточными белками. На рубеже 1970–80-х годов им пользовались десятки лабораторий для идентификации самых разных белков, а сам изобретатель с его помощью быстро охарактеризовал белковые продукты генов uvrA, uvrB иuvrC и показал, что они образуют комплекс, который назвали эксцинуклеазой (Excinuclease) – он был способен вырезать (англ. excise) кусок ДНК размером 13 пар нуклеотидов вокруг тиминового димера. От этого весь механизм получил название эксцизионной репарации нуклеотидов (Nucleotide excision repair). Дальнейшие исследования позволили установить, что после вырезания фрагмента, содержащего повреждение, ДНК-полимераза синтезирует нормальный участок цепи ДНК и процесс репарации завершается ферментом ДНК-лигазой, которая восстанавливает целостность остова ДНК.
Эксцизионная репарация нуклеотидов для жизни в целом гораздо важнее, чем фотореактивация. Например, у человека фотолиазы нет – из всех млекопитающих ее сохранили только сумчатые, а у остальных сохранились гомологи фотолиазы, крипто- хромы, отвечающие за суточные. Поэтому вся репарация вызванных ультрафиолетовым светом повреждений у нас опирается исключительно на эксцизионную репарацию нуклеотидов. Белки этой системы в организме человека не похожи на бактериальные, но принцип работы тот же – вырезать отрезок ДНК и заменить его новым. Дефекты эксцизионной репарации нуклеотидов вызывают тяжелейшее наследственное заболевание – пигментную ксеродерму, при которой малейшее пребывание на солнце приводит к ожогам, и за несколько лет жизни развивается рак кожи. Для этого заболевания очень характерен рак кончика языка – человек на свету облизывает пересохшие губы, и этих нескольких секунд облучения достаточно, чтобы в ДНК возникло столько повреждений, что они в отсутствие репарации вызывают мутации и рак. Еще более важно то, что фотореактивация – процесс ,специфичный только для тиминовых димеров, другие повреждения ею не исправляются, а вот эксцизионная репарация нуклеотидов универсальна и помогает бороться с огромным числом самых разнообразных повреждений ДНК, например, с теми, что вызываются канцерогенами в табачном дыме.
Эксцизионная репарация нуклеотидов исправляет до 10 % всех повреждений, возникающих в ДНК человека. При ее некомпетентности или недостаточности подключаются другие механизмы восстановления ДНК, такие как мисматч-репарация (DNA mismatch repair, от английского слова mismatch – неправильная, неподходящая пара, мезальянс). Аналогом этого названия является термин «репарация гетеродуплексов», «репарация неканонических пар оснований». Это система, которая исправляет ошибки ДНК-полимераз, если те включают в ДНК при синтезе не те нуклеотиды, что нужно, – образуют не пары A:T и G:C, а что-то другое, например G:T. Такое случается редко, но всё же случается, потому что ни один фермент не работает со стопроцентной точностью. Системой распознавания неправильно включенного нуклеотида являются другие ферменты. Помимо этого, важно понимать, что могут быть не поврежденные, а нормальные нуклеотиды, просто не подходящие друг другу по паре оснований. И для этого в организме также существуют специфические ферменты.
Многие бактерии маркируют материнскую цепь при помощи метильных групп, которые специальный фермент, ДНК-метилаза Dam, вводит в основания аденина, находящиеся в последовательностях -GATC-. Таким образом, сразу после синтеза ДНК эта последовательность на протяжении нескольких минут остается полуметилированной – то есть несет метильные группы в материнской цепи и не содержит их во вновь синтезированной дочерней цепи. Этого времени системе мисматч-репарации достаточно для того, чтобы сработать. В организме человека механизм, различающий материнскую и дочернюю цепь, другой и более сложный, основанный на асимметричном связывании некоторых белков при репликации, – но он всё равно существует, мисматч-репарация без такого механизма работать не может.
После маркировки цепей метильными группами образуются дуплексы между цепочками ДНК бактериофагов, отличающихся на один нуклеотид. Это позволило изучать дальнейшие процессы в неправильных парах нуклеотидов и с изолированными белками системы репарации, и в клетках бактерий. Сразу после репликации с полуметилированными последовательностями -GATC- связывается белок MutH. Одновременно с неправильной парой нуклеотидов связываются две молекулы белка MutS. Две молекулы белка оказались очень похожими на сложенные в молитве ладони, между которыми зажата ДНК. Когда расстояние между MutH и димером MutS позволяет им взаимодействовать (в чем им помогает третий член системы, MutL), белок MutH превращается в эндонуклеазу, которая расщепляет неметилированную цепь в последовательности -GATC-. Начиная с этого разрыва, дочерняя цепь ДНК затем удаляется в направлении связанного белка MutS. Достигнув неправильной пары оснований, разрушение ДНК останавливается, после чего недостающий участок ДНК вновь синтезируется.
Полом Модричем были открыты основные принципы мисматч-репарации и у бактерий, и у человека [6]. Система мисматч-репарации в организме человека очень похожа на бактериальную, за исключением принципа определения материнской и дочерней цепи. Мутации в генах, ответственных за мисматч-репарацию, приводят к развитию наследственного рака кишечника и служат самой распространенной причиной этого заболевания.
Самой важной системой репарации являлется эксцизионная репарация оснований. Она устраняет подавляющее большинство всех повреждений. К ним относятся как раз те, которые неизбежно возникают в ДНК под действием воды и кислорода, но и многие другие повреждения тоже ею исправляются. Если поломки в других системах репарации вызывают тяжелые заболевания, неисправность эксцизионной репарации оснований у человека, за редкими исключениями, в заболеваниях не проявляется – эмбрионы гибнут на самых ранних стадиях.
Открытие эксцизионной репарации оснований Томас Линдаль [4, 5] связывает с исследованиями химической реактивности ДНК, к чему его вдохновила знаменитая «Белая книга» – переведенная на английский язык монография «Органическая химия нуклеиновых кислот» академика Н. К. Кочеткова с соавторами [1]. Ранние представления о ДНК, как химически устойчивой молекуле, которая лишь изредка повреждается под влиянием ультрафиолета, радиации или химических мутагенов, в корне неверно – ДНК в водной среде повреждается постоянно. Выбрав две простых и легко идущих химических реакции – превращение цитозина в урацил (который в норме встречается в РНК, но не в ДНК) и апуринизацию (отщепление от ДНК аденина или гуанина), – Линдаль быстро показал, что они протекают и в изолированной ДНК, и в живой клетке. Более того, получив ДНК, в которой часть цитозина была заменена на урацил, он обнаружил и фермент, который удалял урацил в виде свободного основания – урацил-ДНК-гликозилазу (Uracil DNA glycosylases) – и открыл новый вид репарации.
По пути эксцизионной репарации оснований происходит репарация небольших поврежденных оснований и апуринизированных нуклеотидов, которые не вносят значительных искажений в структуру ДНК и поэтому не узнаются системой эксцизионной репарации нуклеотидов. Сначала поврежденное основание узнается одним из ферментов, относящимся к классу ДНК-гликозилаз (DNA glycosylase), которые выщепляют его из ДНК. ДНК-гликозилазы обладают групповой спе- цифичностью – некоторые удаляют из ДНК только окисленные пуриновые основания, другие – окисленные пиримидины, третьи – алкилированные основания, четвертые – урацил и т. п. После этого фермент АП-эндонуклеаза разрывает ДНК рядом с повреждением, ДНК-полимераза встраивает один (так называемая «короткозаплаточная репарация») или несколько нуклеотидов («длиннозаплаточная репарация»), и репарация завершается ДНК-лигазой. В процессе эксцизионной репарации оснований участвуют еще несколько белков, но они играют вспомогательную роль.
Эксцизионная репарация оснований используется не только для восстановления ДНК, но и в других процессах. Например, ту же урацил-ДНК-гликозилазу клетки человека используют для борьбы с вирусами, в частности с ВИЧ. Существует специальный фермент APOBEC [8], который в вирусной ДНК массово превращает цитозин в урацил, а урацил-ДНК-гликозилаза потом такую ДНК расщепляет. Иммунный ответ также требует участия урацил-ДНК-гликозилазы, которая в этом случае отвечает за генерацию разнообразия антител. Эксцизионная репарация оснований лежит в основе эпигенетических процессов – направленной модификации ДНК, которая регулирует активность генов. В раковых клетках некоторые пути репарации выключены, и ингибиторы оставшихся путей, главным образом эксцизионной репарации оснований, сейчас рассматриваются как новые многообещающие лекарства в онкологии.
В России основные исследования репарации ДНК ведутся в нескольких лабораториях Института химической биологии и фундаментальной медицины СО РАН в Новосибирске; есть группы, работающие в этом направлении в МГУ, Институте молекулярной генетики РАН, Институте цитологии РАН в Санкт-Петербурге, Петербургском институте ядерной физики.
Помимо описанных способов репарации ДНК, существуют и уже описанные механизмы ее восстановления, такие как рекомбинационная репарация (Homologous recombination), когда для восстановления правильной последовательности ДНК используется ее копия с другой хромосомы, и воссоединение негомологичных концов (Microhomology-mediated end joining), когда часть ДНК теряется, но это часто неважно, потому что она приходится на некодирующие области. Оба этих вида репарации используются, когда нужно исправить двуцепочечный разрыв ДНК. Есть системы толерантности к повреждению (Translesion synthesis), когда клетка может функционировать и даже делиться, несмотря на то, что с ее геномом не всё в порядке. Есть клеточные системы ответа на повреждение (DNA damage response), которые определяют, как клетке вести себя и функционировать в случае повреждения ее ДНК: делиться, остановить деление и попытаться отрепарировать повреждение, погибнуть или использовать еще какой-нибудь, неизвестный в настоящее время механизм саморегуляции. За исследование последней системы в 2015 году Стефан Эллидж (Stephen Elledge) и Эвелин Виткин (Evelyn M. Witkin) получили Ласкеровскую премию (Lasker Award). Эвелин Виткин открыла первую систему координированного клеточного ответа на повреждение ДНК – SOS-ответ.
Таким образом, все химические реакции, происходящие на уровне ДНК бактерии, свойствены и клеткам человеческого организма, за малым исключением или с некоторыми вариациями. Однако голографичность энзимных механизмов и репаративных процессов ДНК бактерии в общем и целом схожи с химическими процессами, протекающими в клетках макроорганизма, что делает задачу изучения химических процессов репарации ДНК микросистем еще более актуальной.
Молекулярная биология клетки — Том 1 — Албертс Б., Брей Д., Льюис Дж., Рэфф М., Робертс К., Уотсон Дж. 1994
Молекулярная организация клеток
Основные генетические механизмы
Механизмы репликации ДНК
Живые организмы должны не только поддерживать целостность нуклеотидных последовательностей ДНК путем ее репарации, но еще и очень точно воспроизводить свою ДНК перед каждым клеточным делением. При репликации ДНК скорость полимеризации колеблется в пределах от 500 нуклеотидов в 1 с у бактерий приблизительно до 50 нуклеотидов у млекопитающих. Ясно, что ферменты, катализирующие процесс репликации, должны работать и точно, и быстро. Быстрота и точность достигаются с помощью особого мультиферментного комплекса, направляющего процесс репликации. Этот комплекс, состоящий из нескольких различных белков, представляет собой сложный и совершенный «аппарат репликации».
5.3.1. Репликация ДНК, как и ее репарация, основана на комплементарном спаривании оснований [25]
Матричная активность ДНК проявляется в том, что ее нуклеотидная последовательность копируется (целиком или частично) путем комплементарного спаривания оснований (А с Т или G с С) в виде комплементарной последовательности нуклеотидов ДНК или РНК. Этот процесс предполагает узнавание каждого нуклеотида в ДНК свободным (неполимеризованным) комплементарным нуклеотидом и обязательное разделение (хотя бы на время) двух цепей ДНК, с тем чтобы в каждом основании группы, играющие роль доноров и акцепторов при образовании водородных связей, оказались доступными для комплементарного спаривания. Таким образом поступающие одиночные нуклеотиды выстраиваются в определенном порядке вдоль матричной цепи ДНК для ферментативной полимеризации, продуктом которой является новая полинуклеотидная цепь. В 1957 г. был открыт первый фермент, катализирующий процесс полимеризации нуклеотидов; он был назван ДНК-полимеразой. Было показано, что субстратами ДНК-полимеразы служат дезоксирибонуклеозидтрифосфаты, полимеризующиеся на одно-цепочечной ДНК-матрице (двухступенчатый механизм этой полимеризации представлен на рис. 5-34 в связи с обсуждением процесса репарации ДНК). Позже была выделена и РНК-полимераза, для которой субстратами служат рибонуклеозидтрифосфаты.
Во время репликации ДНК каждая из двух ее старых цепей служит матрицей для образования новой цепи. Поэтому чрезвычайно длинная нуклеотидная последовательность клеточной ДНК реплицируется, как это принято называть, «полуконсервативно» и каждая из двух дочерних клеток получает при клеточном делении новую двойную спираль ДНК, состоящую из одной старой и одной новой цепи (см. рис. 3-11).
5-23
5.3.2. Репликационная вилка асимметрична [26]
Исследования, проведенные в начале 1960-х годов на реплицирующихся хромосомах, в которые в качестве импульсной метки вводили радиоактивный предшественник ДНК 3Н-тимидин, выявили особую четко ограниченную область репликации, перемещающуюся вдоль родительской спирали ДНК. Эта активная область из-за своей Y-образной формы была названа репликационной вилкой. Именно в ней с помощью мультиферментного комплекса, содержащего ДНК-полимеразу, синтезируются дочерние молекулы ДНК.
В то время казалось вполне вероятным, что простейший механизм репликации ДНК заключается в непрерывном росте обеих новых цепей нуклеотид за нуклеотидом по мере перемещения репликационной вилки от одного конца молекулы ДНК к другому. Однако, поскольку две цепи в спирали ДНК антипараллельны, одна из дочерних цепей должна расти в направлении 5′ → 3′, а другая — в направлении 3′ → 5′. В таком случае репликационной вилке потребовалось бы две разные ДНК-полимеразы. Одна из них наращивала бы цепь в направлении 5′ → 3′ (рис. 5-34); при этом каждый поступающий мономер (дезоксирибонуклеозидтрифосфат) приносит с собой необходимую для его присоединения к цепи энергию (ее носителем является трифосфатная группа). Другая ДНК-полимераза, перемещающаяся в направлении 3′ → 5′, должна катализировать «рост с головы»; в этом случае энергию, необходимую для присоединения каждого очередного нуклеотида, должен нести конец растущей цепи ДНК. В действительности такой (3′ → 5′) ДНК-полимеразы не существует (рис. 5-38), хотя биохимикам известны некоторые другие процессы полимеризации, протекающие по типу «роста с головы» (см. рис. 2-34), Каким же образом происходит рост цепи в направлении 3′ → 5′? Возможный ответ на этот вопрос подсказали в конце 1960-х годов эксперименты с радиоактивно меченными предшественниками ДНК. Если растущие клетки получают всего на несколько секунд высокорадиоактивный 3Н-тимидин, то метка включается лишь в ДНК, синтезированную в самый последний момент, т. е. в ту ее часть, которая следует непосредственно за репликационной вилкой. Этим методом избирательного введения метки было выявлено, что при репликации бактериальной ДНК в области репликационной вилки образуются и какое-то время существуют фрагменты, насчитывающие от 1000 до 2000 нуклеотидов (впоследствии за ними закрепилось название «фрагменты Оказаки»; у эукариот они гораздо короче: от 100 до 200 нуклеотидов). Несколько позже было показано, что синтез этих фрагментов ДНК идет только в направлении 5′ → 3′; синтезированные фрагменты соединяются затем в длинные цепи ДНК под действием того же фермента, который сшивает разрывы в спирали ДНК во время ее репарации, т.е. под действием ДНК-лигазы (см. рис. 5-35).

Рис. 5-38. На первый взгляд простейшим механизмом репликации ДНК представляется механизм, изображенный на этой (неверной!) схеме. Обе дочерние цепи должны были бы при этом расти непрерывно за счет присоединения нуклеотидов соответственно в 5′ → 3′ — направлении (на рисунке — внизу) и 3′ → 5′ — направлении (на рисунке — вверху). Однако фермента, который бы катализировал присоединение нуклеотидов в направлении 3′ → 5′, не существует.

Рис. 5-39. Строение репликационной вилки. Обе дочерние цепи строятся в направлении 5′ → 3′. Для этого отстающая цепь ДНК должна синтезироваться в виде ряда коротких фрагментов (фрагменты Оказаки).
Репликационная вилка асимметрична (рис. 5-39). Из двух синтезируемых дочерних цепей ДНК одна строится непрерывной, а другая прерывистой. Первую называют ведущей (или лидирующей), а вторую — отстающей. Наращивание второй цепи отстает, потому что образование каждого фрагмента Оказаки оказывается возможным лишь после того, как продвижение ведущей цепи откроет соответствующий участок матрицы. Хотя в целом вся эта цепь строится в направлении 3′ → 5′, каждый из ее фрагментов синтезируется в направлении 5′ → 3′. Благодаря тому что ДНК на отстающей части вилки строится при помощи механизма, работающего прерывисто по типу «шитья назад иголкой» (backstitching), в репликационной вилке не требуется никакого другого фермента, кроме (5′ → 3′)-ДНК-полимеразы.
5-24
5.3.3. Высокая точность репликации ДНК предполагает наличие механизма, осуществляющего коррекцию [27]
Точность копирования при репликации ДНК столь велика, что в среднем на каждые 1-109 комплементарных пар, образующихся в процессе воспроизведения генома млекопитающих, насчитывающего 3-104 пар оснований (см. разд. 9.1.3), приходится приблизительно одна ошибка. Точность эта значительно превосходит ту, какую следует ожидать, учитывая, что во время репликации образуются не только обычные комплементарные пары оснований. В нормальной ДНК возникают на короткое время с частотой 10-4-10-5 редкие таутомерные формы всех четырех ее оснований. Эти формы образуют неправильные пары. Так, редкая таутомерная форма С спаривается с А вместо G, в результате чего возникает мутация (рис. 5-40). Таким образом высокая точность репликации ДНК определяется наличием механизмов, осуществляющих коррекцию, т. е. устраняющих подобные ошибки.

Рис. 5-40. Пример возникновения при репликации ДНК неправильной пары оснований: находясь в термодинамически невыгодной таутомерной форме, цитозин легко образует водородные связи с аденином.

Рис. 5-41. Схема, поясняющая, как протекает процесс коррекции (устранение ошибок) при синтезе ДНК, катализируемом ДНК-полимеразами у бактерий. Предполагается, что аналогичный механизм коррекции действует и в эукариотических клетках.
Один из важных механизмов коррекции зависит от особых свойств ДНК-полимеразы. В отличие от РНК-полимераз ДНК-полимеразы не могут начать синтез новой полинуклеотидной цепи, просто связав друг с другом 3′-ОН- конец какой-либо полинуклеотидной цепи, которая должна быть спарена с матричной цепью ДНК; ДНК-полимеразы способны только добавлять новые нуклеотиды к уже имеющемуся 3′-ОН-концу полинуклеотидной цепи (см. рис. 534). Эту предобразованную цепь, к которой добавляются нуклеотиды, называют затравкой или праймером. Молекулы ДНК с затравкой, у которой 3′-ОН-конец не спарен, не могут служить матрицами. Бактериальные ДНК-полимеразы способны, однако, с ними работать. Вступив в контакт с такими молекулами ДНК, они используют присущую им (3′ → 5′)-экзонуклеазную активность и отщепляют (путем гидролиза) любые неспаренные нуклеотиды на затравочном конце. Отщепляется ровно столько нуклеотидов, сколько требуется для того, чтобы у затравки появился спаренный конец и образовалась активная матрица. Действуя таким образом, ДНК-полимераза выступает в роли «самокорректирующего» фермента: она устраняет свои собственные ошибки, возникающие в процессе полимеризации. Рис. 5-41 поясняет, как этот тип коррекции может использоваться для удаления неправильных пар С—А, образуемых редкой таутомерной формой цитозина.
Потребность в правильно спаренном конце как раз и наделяет ДНК- полимеразу способностью исправлять свои собственные ошибки. Такой фермент, очевидно, мог бы начать синтез ДНК при полном отсутствии затравки, только утратив способность различать спаренный и неспаренный концы. В то же время РНК-полимеразы, участвующие в транскрипции генов (см. разд. 5.1.1), судя по всему не нуждаются в самокоррекции, потому что ошибки транскрипции не передаются следующему поколению и случайно возникшие дефектные молекулы особой роли не играют. РНК-полимеразы могут начинать синтез новых полинуклеотидных цепей в отсутствие затравки, причем ошибки встречаются с частотой 10-4 как при синтезе РНК, так и при трансляции, т.е. при переводе нуклеотидных последовательностей мРНК в аминокислотные последовательности белков.
5-22
5.3.4. Репликация ДНК в направлении 5’ → 3’ обеспечивает эффективную коррекцию
Весьма вероятно, что однонаправленность репликации ДНК (5′ → 3′) объясняется высокими требованиями к точности процесса. Если бы существовала ДНК-полимераза, присоединяющая дезоксирибонуклеозидтрифосфаты к синтезируемой полинуклеотидной цепи таким образом, что эта цепь росла в направлении 3′ → 5′, то активирующую трифосфатную группировку нес бы растущий 5′-конец цепи, а не поступающий мононуклеотид. В этом случае ошибки полимеризации не могли бы устраняться простым гидролизом, потому что появление свободного 5′-конца немедленно обрывало бы синтез ДНК. Ясно, что основание, только что неправильно спарившееся на 3′-конце, устранить гораздо легче, чем такое же основание, присоединившееся к 5′-концу цепи ДНК. Поэтому, хотя механизм репликации ДНК, изображенный на рис. 5-39, кажется на первый взгляд значительно более сложным и громоздким, чем неверный гипотетический механизм, представленный на рис. 5-38, этот реально функционирующий механизм способен обеспечить гораздо большую точность именно в силу того, что синтез ДНК идет здесь только в направлении 5′ → 3′.
5-25
5.3.5. Для синтеза коротких затравочных молекул на матрице отстающей цепи требуется особый фермент [28]
С того момента, как возникла репликационная вилка, для ДНК-полимеразы, синтезирующей ведущую цепь, всегда есть спаренный 3′-конец, необходимый ей для того, чтобы начать синтез новой цепи. Иначе обстоит дело с ДНК-полимеразой, ответственной за синтез отстающей цепи. Ей требуется всего каких-нибудь 4 с для того, чтобы синтезировать один короткий фрагмент ДНК, после чего она должна переключиться на синтез совсем другого фермента на новом участке матричной цепи, расположенной на некотором расстоянии от первого (см. рис. 5-39). Для этого ей всякий раз нужна затравка со спаренным 3′-концом, а следовательно, нужен и механизм, способный производить такие затравки. В этот механизм входит фермент, называемый ДНК-праймазой. Она синтезирует из рибонуклеозидтрифосфатов короткие РНК-затравки (праймеры), состоящие у эукариот примерно из 10 нуклеотидов (рис. 5-42). Эти затравки синтезируются с определенными интервалами на матрице для отстающей цепи; здесь их наращивает ДНК-полимераза, начиная, таким образом, всякий раз новый фрагмент Оказаки. Молекула ДНК-полимеразы продолжает это наращивание до тех пор, пока она не достигнет РНК-затравки, присоединенной к 5′-концу предыдущего фрагмента ДНК. Чтобы обеспечить образование непрерывной цепи ДНК из многих таких фрагментов, в действие вступает особая система репарации ДНК, быстро удаляющая РНК-затравку и заменяющая ее на ДНК. Завершает процесс ДНК-лигаза, соединяющая 3′-конец нового фрагмента ДНК с 5′-концом предыдущего фрагмента (рис. 5-43).
Почему предпочтение отдается удаляемой РНК-затравке, а не ДНК-затравке, которую не требовалось бы удалять? Выше мы отмечали, что самокорректирующая полимераза не способна начинать синтез полинуклеотидных цепей de novo; это предполагает и обратное утверждение: тот фермент, который начинает синтез цепей de novo, к эффективной самокоррекции не способен. Значит, любой фермент, катализирующий инициацию синтеза фрагментов Оказаки, неизбежно создал бы не слишком точную копию (не менее 1 ошибки на 105). Это означало бы колоссальное увеличение частоты мутаций даже при том, что количество таких копий, сохранившееся в конечном продукте, составляло бы не более 5% всего генома (например, 10 нуклеотидов во фрагменте, состоящем из 200 нуклеотидов). Естественно думать поэтому, что выдвижение РНК, а не ДНК на роль затравки обеспечивало важное преимущество, поскольку рибонуклеотиды автоматически метят такие последовательности, как «плохие копии», которые должны быть удалены.

Рис. 5-42. Схема реакции, катализируемой праймазой — ферментом, синтезирующим короткие РНК-затравки в отстающей цепи ДНК. В отличие от ДНК-полимеразы этот фермент способен начинать синтез новой полинуклеотидной цепи с соединения двух нуклеозидтрифосфатов. Образовав короткий полинуклеотид, праймаза прекращает работу. Теперь к свободному 3′-концу может добавлять нуклеотиды ДНК-полимераза.
5-26
5-33
5.3.6. Особые белки способствуют расплетанию двойной спирали ДНК перед репликационной вилкой [29]
Двойная спираль ДНК должна расплетаться по ходу продвижения репликационной вилки, для того чтобы поступающие дезоксирибонуклеозидтрифосфаты могли спариваться с родительской матричной цепью. Однако в обычных условиях двойная спираль ДНК весьма стабильна; спаренные основания соединены столь прочно, что для разделения двух цепей ДНК в пробирке требуются температуры, приближающиеся к точке кипения воды (90°С). По этой причине большинство ДНК-полимераз может копировать лишь ту молекулу ДНК, у которой матричная цепь уже отделилась от другой цепи. Для того чтобы двойная спираль ДНК раскрылась и соответствующая матричная цепь стала доступной для ДНК-полимеразы, необходимы особые белки. Они бывают двух типов.
ДНК-геликазы были впервые выделены как белки, которые, присоединяясь к одиночной цепи ДНК, катализируют гидролиз АТР. Как уже отмечалось в гл. 3, гидролиз АТР может циклическим образом изменять форму молекулы белка, вследствие чего белок будет производить механическую работу (см. разд. 3.4.11). Именно этот принцип лежит в основе быстрого перемещения ДНК-геликаз по одиночной цепи ДНК. Встречая на своем пути участок двойной спирали, эти ферменты продолжают двигаться вдоль своей цепи и тем самым расплетают двойную спираль (рис. 5-44). Расплетание ДНК-спирали в области репликационной вилки, вероятно, осуществляется двумя совместно действующими ДНК- геликазами, одна из которых перемещается по ведущей цепи, а другая — по отстающей. Ясно, что две эти геликазы должны двигаться вдоль одиночных цепей ДНК в противоположных направлениях, т. е. это должны быть разные ферменты. Действительно, оба указанных типа ДНК- геликаз удалось обнаружить. При этом исследования на бактериях показали, что главную роль играет ДНК-геликаза отстающей цепи. Причины этого мы обсудим ниже.
Белки, дестабилизирующие спираль (их называют также белками, связывающими одноцепочечную ДНК или SSB-белками), связываются с одиночными цепями ДНК, не закрывая оснований, т. е. оставляя их доступными для спаривания. Сами они не способны расплетать длинные молекулы ДНК, но, присоединяясь к одиночным цепям ДНК, они тем самым способствуют любому процессу расплетания спирали; они, например, помогают ДНК-геликазе расплетать двойную спираль в репликационной вилке. На матрице отстающей цепи SSB-белки кооперативным образом связываются с одноцепочечными участками ДНК и предотвращают здесь образование «шпилек», небольших двухспиральных структур, которые могли бы помешать синтезу ДНК, осуществляемому ДНК-полимеразой (рис. 5-45).

Рис. 5-43. Отдельные этапы синтеза каждого из фрагментов отстающей цепи ДНК. У эукариот РНК-затравки синтезируются в отстающей цепи с интервалами приблизительно в 200 нуклеотидов и каждая из них состоит из 10 нуклеотидов.
5.3.7. Белки в репликационной вилке действуют кооперативно, образуя «репликационную машину» [30]
До сих пор мы говорили о репликации ДНК так, как если бы она осуществлялась смесью репликационных белков, действующих независимо друг от друга. Между тем в действительности большая часть этих белков объединена в крупный мультиферментный комплекс, быстро движущийся вдоль ДНК. Этот комплекс — нечто вроде крошечной «швейной машины»: «деталями» его служат отдельные белки, а источником энергии — реакция гидролиза нуклеозидтрифосфата. Комплекс изучен достаточно хорошо только у бактерий Е. coli и у некоторых вирусов, но есть все основания считать, что очень похожий механизм действует и у эукариот (см. разд. 9.3.3).
Схема на рис. 5-46, где подробно изображена репликационная вилка, позволяет судить о том, как работают отдельные части такой «репликационной машины». В области вилки действуют две идентичные ДНК-полимеразы — на ведущей и на отстающей цепи. Спираль ДНК расплетается в результате совместного действия ДНК-полимеразы, работающей на ведущей цепи, и ДНК-геликазы, движущейся вдоль отстающей цепи; этому процессу способствуют кооперативно связывающиеся молекулы дестабилизирующегося белка. В то время как на ведущей цепи ДНК- полимераза работает непрерывно, на отстающей цепи фермент через определенные интервалы прерывает и вновь возобновляет свою работу, используя для полимеризации короткие РНК-затравки, синтезируемые ДНК-праймазой.
Эффективность репликации сильно возрастает вследствие тесного объединения всех этих белковых компонентов. Молекула праймазы непосредственно сцеплена с ДНК-геликазой, образуя вместе с нею на отстающей цепи структуру, называемую праймосомой, которая движется с репликационной вилкой и по ходу своего движения синтезирует РНК-затравки. Молекула ДНК-полимеразы, работающая на отстающей цепи, также движется совместно с остальными белками, синтезируя ряд новых фрагментов Оказаки; ради этого, как полагают, цепь ДНК, которая служит для нее матрицей, складывается сама на себя, как это показано на рис. 5-47. Репликационные вилки оказываются, таким образом, объединены в одну крупную структуру (с общей массой > 106 дальтон), быстро перемещающуюся вдоль ДНК и обеспечивающую возможность координированного и эффективного синтеза ДНК на обет ветвях вилки.

Рис. 5-44. Действие ДНК-геликаз. Небольшой фрагмент ДНК присоединен путем отжига к длинной одноцепочечной ДНК, так что образовался короткий участок двойной спирали. Эта спираль расплетается по мере того, как геликаза движется вдоль одиночной цепи ДНК, катализируя реакцию, для которой требуется наряду с ферментом и АТР. Источником энергии для движения геликазы служит гидролиз АТР (см. рис. 3-63).

Рис. 5-45. Влияние дестабилизирующих белков на структуру одно-цепочечной ДНК. Поскольку каждая белковая молекула предпочитает связываться с другой, уже связавшейся ранее молекулой (так называемое кооперативное связывание), эти белки образуют длинные кластеры, выпрямляющие матричные пени ДНК и облегчающие процесс полимеризации. Структуры в форме «шпильки», возникающие в свободной одноцепочечной ДНК, образуются путем случайного спаривания оснований в коротких участках, содержащих взаимно комплементарные последовательности нуклеотидов, они напоминают короткие спирали, возникающие во всех молекулах РНК.

Рис. 5-46. Главные типы белков, действующих в области репликационной вилки (схема показывает их локализацию на ДНК). Комплекс ДНК-праймазы и ДНК-геликазы на отстающей цепи ДНК известен под названием праймосомы.
Позади «репликационной машины» по ходу ее движения остается на отстающей цепи ряд несшитых фрагментов Оказаки, все еще содержащих на своем 5′-конце РНК-затравки, необходимые для инициации на синтеза. Эти РНК-затравки должны быть удалены, а фрагменты сшиты при помощи репарирующих ферментов, работающих позади репликационной вилки (см. рис. 5-43).

Рис. 5-47. Схема, иллюстрирующая современные представления о расположении репликационных белков в движущейся репликационной вилке. Вместо двумерной структуры, изображенной на рис. 5-46, здесь показано, как ДНК на отстающей цепи складывается, в результате чего возникает комплекс из двух ДНК-полимераз — ведущей и отстающей цепи. Кроме того, благодаря складыванию 3′-конец каждого завершенного фрагмента Оказаки оказывается рядом со стартовым участком следующего такого фрагмента (ср. с рис. 5-46). Находясь в тесном контакте с остальными репликационными белками, молекула ДНК-полимеразы отстающей цепи может непрерывно работать на одной и той же репликационной вилке, отделяясь от готового фрагмента ДНК, она переходит к ближайшей новой РНК-затравке, чтобы начать синтез следующего фрагмента. Обратите внимание, что на этой схеме одна из дочерних спиралей ДНК направлена вправо и вниз, а вторая влево и вверх.
5.3.8. Ошибки при репликации ДНК в бактериальных клетках устраняются особой корректирующей системой, распознающей неправильное спаривание оснований
У таких бактерий, как Е. coli, деление происходит каждые 30 мин, поэтому у них сравнительно легко выявить в большой популяции клеток редкие экземпляры с измененными признаками. Выделен, например, класс мутантов, характеризующихся резким повышением частоты спонтанных мутаций, что связано с присутствием в его клетках специфических генов-мутаторов. Известен ген-мутатор, кодирующий дефектную форму 3′ → 5′-корректирующей экзонуклеазы, представляющей собой субъединицу ДНК-полимеразы (см. разд. 5.3.3). Если дефект затрагивает этот белок, то ДНК-полимераза утрачивает способность эффективно осуществлять коррекцию и в ДНК накапливается много ошибок, которые при нормальной репликации были бы устранены.
Изучение тех мутантов Е. coli, у которых имеются гены-мутаторы, выявило еще одну систему, в норме устраняющую ошибки репликации, не улавливаемые корректирующей экзонуклеазой. Эта система коррекции неправильного спаривания (mismatch proofreading sistem), называемая также системой исправления ошибок спаривания (mismatch repair system), отличается от ранее рассмотренных систем репарации ДНК тем, что она не зависит от присутствия в ДНК аномальных нуклеотидов, которые должны быть распознаны и удалены («вырезаны»). Она выявляет деформации на внешней стороне спирали, вызванные плохой пригонкой обычных, но некомплементарных оснований. Если бы эта корректирующая система просто распознавала ошибки спаривания в реплицировавшейся ДНК и удаляла без выбора один из двух неправильно спарившихся нуклеотидов, то в половине случаев она бы сама совершала ошибку, «исправляя» не новосинтезированную, а матричную цепь, так что в среднем частота ошибок оставалась бы прежней. Для эффективной коррекции система должна уметь различать неправильно спаривающиеся нуклеотиды и избирательно удалять такие нуклеотиды только из новой цепи (т.е. устранять именно ошибки репликации).
В клетках Е. coli процесс распознавания связан с метилированием определенных остатков аденина в ДНК. Метальные группы присоединяются ко всем остаткам А в последовательности GATC, но лишь спустя некоторое время после того, как А включится в новосинтезированную цепь ДНК. Новые цепи отличаются от старых тем, что только в них сразу же за репликационной вилкой могут находиться еще не метилированные последовательности GATC. Коррекция неправильного спаривания осуществляется крупным мультиферментным комплексом, сканирующим каждую из двух цепей двойной спирали ДНК. Этот комплекс удаляет только неправильно присоединенные нуклеотиды, но делает это лишь после того, как на той же цепи обнаружится и неметилированная последовательность GATC. Поэтому нуклеотиды удаляются только из новой цепи, т. е. устраняются ошибки репликации (рис. 5-48).

Рис. 5-48. Схема эксперимента, иллюстрирующего работу системы коррекции неправильного спаривания, устраняющей у бактерий ошибки репликации ДНК. Особый белковый комплекс удаляет неспаренные нуклеотиды из вновь синтезируемой цепи ДНК позади репликационной вилки, этот репарирующий комплекс узнает новую цепь ДНК по обнаруживаемым в ней неметилированным последовательностям GATC. На схеме представлены три молекулы ДНК с одной и той же «неправильной» парой нуклеотидов, но при этом в одной молекуле (А) метилированные последовательности GATC встречаются в обеих цепях, в другой молекуле (Б) таких метилированных последовательностей нет совсем, а в третьей (В) они присутствуют только в одной из цепей. Если воздействовать на эти молекулы ДНК клеточным экстрактом, содержащим корректирующий комплекс, то мы получим представленный здесь результат. Молекула ДНК в правой части рисунка воспроизводит картину, обнаруживаемую непосредственно за репликационной вилкой: нижняя цепь соответствует новой цепи, где метилирование еще не произошло.
В эукариотических клетках не удалось пока выявить ни одного из этих двух механизмов коррекции, обнаруженных у бактерий. Однако степень точности репликации у млекопитающих и у Е. coli приблизительно одинакова, и потому можно думать, что оба описанных типа коррекции существуют и у эукариот. Следует, впрочем, отметить, что в ДНК млекопитающих нет метилированных остатков А, поэтому механизм, который используется системой репарации ошибок спаривания для узнавания новосинтезированной цепи, должен быть в данном случае иным.
5-27
5.3.9. Репликационные вилки возникают в точках начала репликации [32]
И у бактерий, и у млекопитающих образование репликационных вилок начинается с возникновения особой структуры, называемой репликационным глазком (replication bubble). Это небольшой участок, в котором две цепи родительской спирали ДНК отделились одна от другой и были использованы в качестве матриц для синтеза ДНК (рис. 5-49). Для бактерий и некоторых вирусов, размножающихся в эукариотических клетках, удалось показать, что репликационный глазок образуется в тех местах молекулы ДНК, где находятся специфические нуклеотидные последовательности, получившие название точек начала репликации. Эти последовательности состоят приблизительно из 300 нуклеотидов. Предполагают, что аналогичные точки начала репликации существуют и в эукариотических хромосомах, однако надежных доказательств этого пока нет (см. разд. 9.3.2).
Процесс возникновения репликационных вилок удалось в некоторых случаях воспроизвести in vitro. Эти опыты показали, что у бактерий и бактериофагов инициация репликационных вилок начинается так, как это представлено на рис. 5-50. Множество копий инициаторного белка связываются с особыми участками в точке начала репликации, образуя крупный белковый комплекс. Этот комплекс присоединяет затем ДНК-геликазу и помещает ее на свободную одиночную цепь ДНК в прилегающем участке спирали. Присоединяется также ДНК-праймаза, т. е. образуется праймосома, которая, двигаясь от точки начала репликации, синтезирует РНК-затравку, что дает возможность начать синтез первой цепи ДНК. Остальные белки быстро объединяются после этого в два репликационных белковых комплекса, которые теперь движутся от точки начала репликации в противоположных направлениях (см. рис. 5-49); они продолжают синтезировать ДНК до тех пор, пока обе вилки не пройдут путь по матрице до самого конца.
Некоторые дополнительные данные, касающиеся инициации репликационных вилок в хромосомах эукариот, мы обсудим в гл. 9, там, где речь пойдет о клеточном ядре.

Рис. 5-49. Гипотетический механизм образования репликационных вилок в точках начала репликации (см. также рис. 5-50).

Рис. 5-50. Упрощенная схема, иллюстрирующая начальные этапы образования репликационных вилок в точках начала репликации у Е. coli и бактериофага X. Для обнаружения данного механизма потребовались опыты in vitro с использованием смеси высокоочищенных белков.
Последующие этапы приводят (пока не ясным путем) к инициации еще трех цепей ДНК (рис. 5-49). У Е. coli в репликации ДНК роль инициаторного белка играет белок dnaA; а праймосома состоит из белков dnaB (ДНК-геликаза) и dnaG (ДНК-праймаза).
5.3.10. ДНК-топоизомеразы предотвращают спутывание ДНК во время репликации [33]
Изображая спираль ДНК так, как мы это делали до сих пор, т.е. неправильно, в виде плоской «лестницы», мы игнорировали «проблему закручивания» (winding problem). Между тем на каждые 10 пар оснований, образующихся в репликационной вилке, родительская двойная спираль должна совершить один полный оборот вокруг своей оси. Следовательно, для того чтобы репликационная вилка могла продвигаться вперед, вся хромосома впереди нее должна быстро вращаться (рис. 5-51), что для длинных хромосом потребовало бы большой затраты энергии. При репликации ДНК эта проблема решается иначе: путем образования в спирали своего рода «шарнира», особого класса белков, называемых ДНК- топоизомеразами.
ДНК-топоизомераза представляет собой нечто вроде «обратимой нуклеазы». Сначала она разрывает цепь ДНК, а затем ковалентно присоединяется к разорванному концу. Ковалентная связь белок — ДНК обладает довольно значительной энергией, потому что в ней сохраняется энергия разорванной фосфодиэфирной связи. Вследствие этого реакция, приводящая к разрыву цепи, обратима и не требует дополнительной затраты энергии. В этом отношении данный механизм существенно отличается от механизма действия ДНК-лигазы, о котором мы говорили выше (см. рис. 5-35).
Существуют различные типы ДНК-топоизомераз. Топоизомераза типа I разрывает только одну из двух цепей двойной спирали ДНК, что дает возможность двум участкам ДНК по обе стороны от разрыва свободно вращаться относительно друг друга вокруг фосфодиэфирной связи, находящейся напротив разрыва, которая в этом случае выполняет роль упомянутого выше «шарнира» (рис. 5-52). Всякое напряжение в спирали ДНК заставляет ее вращаться в таком направлении, чтобы ослабить это напряжение. Поэтому вращение во время репликации ДНК происходит лишь на коротком отрезке спирали — в той части, которая находится непосредственно перед репликационной вилкой. Аналогичная проблема, возникшая в процессе транскрипции ДНК, решается таким же путем.

Рис. 5-51. «Проблема кручения», возникающая при репликации ДНК. Для того чтобы репликационная вилка (у бактерий) могла продвигаться вперед со скоростью 500 нуклеотидов в 1 с, родительская спираль ДНК перед вилкой должна вращаться со скоростью 50 об/с.

Рис. 5-52. Обратимая реакция, приводящая к появлению разрыва в одной из цепей ДНК. Реакция у эукариот катализируется ДНК-топоизомеразой типа I Ферменты этой группы образуют временную ковалентную связь с ДНК.

Рис. 5-53. Пример реакции разделения двух сцепленных кольцевых молекул ДНК, катализируемой ДНК-топоизомеразой типа II. Действие этих ферментов (в отличие от реакций, катализируемых ДНК-топоизомеразами типа I) сопряжено с гидролизом АТР и некоторые из них способны сообщать спирали ДНК дополнительное напряжение. ДНК-топоизомеразы типа II обнаруживаются и у прокариот, и у эукариот, по всей вероятности, они участвуют во многих реакциях, имеющих отношение к ДНК.
Топоизомераза типа II ковалентно связывается с обеими цепями двойной спирали ДНК и вносит в нее на время двухцепочечный разрыв. Ферменты этого типа активируются под действием тех участков на хромосомах, где перекрестились спирали. Присоединившись к такому перекресту, топоизомераза: 1) обратимо разрывает одну из двух двойных спиралей, создавая тем самым для другой своего рода «ворота», 2) вынуждает вторую двойную спираль пройти через этот разрыв и 3) сшивает обе разорванные цепи, а затем отделяется от ДНК. Действуя подобным образом, топоизомеразы типа II очень быстро разделяют две сцепленные кольцевые молекулы ДНК (рис. 5-53). Точно так же предотвращают они и спутывание молекул ДНК, которое в противном случае неизбежно создавало бы при репликации серьезную проблему. Известны температурочувствительные мутанты дрожжей, вырабатывающие топоизомеразу II, которая при 37°С инактивируется. Если нагреть эти дрожжевые клетки до такой температуры, то их хромосомы в процессе митоза остаются спутанными и не могут разойтись. Насколько необходим для распутывания хромосом такой «инструмент», как топоизомераза II, поймет каждый, кто хоть раз пытался распутать безнадежно запутавшуюся леску, не имея под рукой ножниц.
5-28
5.3.11. Репликация ДНК у эукариот и прокариот в основных чертах сходна [24]
Почти все, что мы знаем о репликации ДНК, удалось выяснить в опытах с очищенными мультиферментными системами бактерий и бактериофагов, способными осуществлять репликацию ДНК in vitro. Получение таких систем в 1970-х годах заметно облегчилось после того, как удалось выделить мутанты по целому ряду различных генов, ответственных за репликацию, которые можно было использовать для идентификации и очистки соответствующих белков (рис. 5-54).
У эукариот энзимология репликации ДНК пока еще детально не изучена, главным образом потому, что получать здесь необходимые мутантные формы гораздо труднее. Однако схема репликации у прокариот и эукариот в основных чертах, включая геометрию репликационной вилки и потребность в РНК-затравке, по-видимому, одинакова. Главное различие заключается в том, что у эукариот ДНК реплицируется не как таковая, а в виде хроматина, в котором она прочно связана с белками, принадлежащими к классу гистонов. В гл. 8 мы узнаем, что гистоны образуют комплексы в форме дисков, вокруг которых обвивается эукариотическая ДНК, в результате чего возникают регулярно повторяющиеся структуры, называемые нуклеосомами. Нуклеосомы располагаются вдоль молекулы ДНК с интервалами 200 пар оснований. Быть может, именно этим объясняется тот факт, что новые фрагменты отстающей цепи ДНК закладываются у эукариот с интервалами в 10 раз более короткими (от 100 до 200 нуклеотидов), чем у бактерий (от 1000 до 2000 нуклеотидов). Кроме того, если нуклеотиды служат барьерами, на время останавливающими продвижение ДНК-полимеразы, присутствие хроматина (а не голой ДНК) может, вероятно, объяснить и то, что репликационные вилки движутся у эукариот приблизительно в 10 раз медленнее, чем у бактерий.

Рис. 5-54. Получение у бактерий и бактериофагов мутантов с различными нарушениями репликации ДНК открыло возможности для выявления и очистки ферментов, выполняющих какую-либо еще не известную функцию, необходимую для репликации ДНК у прокариот. Использованные здесь температурочувствительные мутанты принадлежат к так называемым условным мутантам, обычно их фермент нормально функционирует при низкой температуре и не работает при высокой. У «безусловных» мутантов с нарушениями репликации синтез ДНК не идет ни при низкой, ни при высокой температуре, и потому эти мутанты обречены на гибель. В модифицированной форме такие «тесты на комплементацию in vitro» полезны также при биохимическом изучении многих других процессов.
Заключение
Самокорректирующая ДНК-полимераза катализирует полимеризацию нуклеотидов на обеих цепях спирали ДНК в направлении 5′ → 3′, копируя матрицу с высокой степенью точности. Поскольку две цепи двойной спирали ДНК аптипараллелъны, в направлении 5′ → 3′ может непрерывно синтезироваться лишь одна из двух цепей (ее называют ведущей). Другая, отстающая цепь синтезируется в виде коротких фрагментов по принципу «шитья назад иголкой». Самокорректирующая ДНК-полимераза не способна начинать синтез новой цепи. Поэтому для закладки фрагментов отстающей цепи ДНК используются короткие молекулы РНК-затравки, которые позже удаляются — их заменяет ДНК.
Процесс репликации ДНК требует совместного действия многих белков. В нем участвуют: 1) ДНК-полимераза и ДНК-праймаза, катализирующие полимеризацию нуклеозидтрифосфатов; 2) ДНК-геликазы и дестабилизирующие белки, помогающие расплести спираль ДНК, которую предстоит копировать; 3) ДНК-лигаза и фермент, разрушающий молекулы РНК-затравки; они нужны для сшивания прерывисто синтезируемых фрагментов отстающей цепи ДНК; 4) ДНК-топоизомеразы, помогающие решить проблемы кручения и спутывания спирали ДНК; 5) инициаторные белки, присоединившиеся к специфическим последовательностям ДНК в точке начала репликации и способствующие образованию здесь новой репликационной вилки. В точке начала репликации к ДНК-матрице сначала присоединяется белковый комплекс, состоящий из ДНК- геликазы и ДНК-праймазы (его называют праймосомой); затем к этому комплексу добавляются другие белки и возникает мультиферментный комплекс — «репликационная машина», которая и осуществляет синтез ДНК.
